python pylab plot normal distribution
Biorąc pod uwagę średnią i wariancję, czy istnieje proste wywołanie funkcji pylab, które wykreśli rozkład normalny?
7 answers
import matplotlib.pyplot as plt
import numpy as np
import matplotlib.mlab as mlab
import math
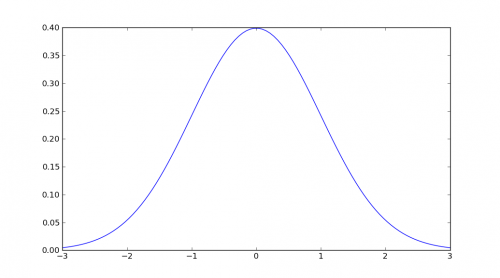
mu = 0
variance = 1
sigma = math.sqrt(variance)
x = np.linspace(mu - 3*sigma, mu + 3*sigma, 100)
plt.plot(x,mlab.normpdf(x, mu, sigma))
plt.show()
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2017-10-15 22:14:51
Nie wydaje mi się, aby istniała funkcja, która robi to wszystko w jednym wywołaniu. Jednak można znaleźć Gaussowską funkcję gęstości prawdopodobieństwa w scipy.stats.
Więc najprostszy sposób mogłem wymyślić jest:
import numpy as np
import matplotlib.pyplot as plt
from scipy.stats import norm
# Plot between -10 and 10 with .001 steps.
x_axis = np.arange(-10, 10, 0.001)
# Mean = 0, SD = 2.
plt.plot(x_axis, norm.pdf(x_axis,0,2))
plt.show()
Źródła:
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2018-07-11 12:24:39
Odpowiedź Unutbu jest poprawna. Ale ponieważ nasza średnia może być większa lub mniejsza niż zero, nadal chciałbym to zmienić:
x = np.linspace(-3 * sigma, 3 * sigma, 100)
Do tego:
x = np.linspace(-3 * sigma + mean, 3 * sigma + mean, 100)
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2018-07-11 11:55:16
Jeśli wolisz używać podejścia krok po kroku, możesz rozważyć rozwiązanie takie jak poniżej
import numpy as np
import matplotlib.pyplot as plt
mean = 0; std = 1; variance = np.square(std)
x = np.arange(-5,5,.01)
f = np.exp(-np.square(x-mean)/2*variance)/(np.sqrt(2*np.pi*variance))
plt.plot(x,f)
plt.ylabel('gaussian distribution')
plt.show()
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2017-12-11 23:18:46
Możesz łatwo uzyskać cdf. so pdf via cdf
import numpy as np
import matplotlib.pyplot as plt
import scipy.interpolate
import scipy.stats
def setGridLine(ax):
#http://jonathansoma.com/lede/data-studio/matplotlib/adding-grid-lines-to-a-matplotlib-chart/
ax.set_axisbelow(True)
ax.minorticks_on()
ax.grid(which='major', linestyle='-', linewidth=0.5, color='grey')
ax.grid(which='minor', linestyle=':', linewidth=0.5, color='#a6a6a6')
ax.tick_params(which='both', # Options for both major and minor ticks
top=False, # turn off top ticks
left=False, # turn off left ticks
right=False, # turn off right ticks
bottom=False) # turn off bottom ticks
data1 = np.random.normal(0,1,1000000)
x=np.sort(data1)
y=np.arange(x.shape[0])/(x.shape[0]+1)
f2 = scipy.interpolate.interp1d(x, y,kind='linear')
x2 = np.linspace(x[0],x[-1],1001)
y2 = f2(x2)
y2b = np.diff(y2)/np.diff(x2)
x2b=(x2[1:]+x2[:-1])/2.
f3 = scipy.interpolate.interp1d(x, y,kind='cubic')
x3 = np.linspace(x[0],x[-1],1001)
y3 = f3(x3)
y3b = np.diff(y3)/np.diff(x3)
x3b=(x3[1:]+x3[:-1])/2.
bins=np.arange(-4,4,0.1)
bins_centers=0.5*(bins[1:]+bins[:-1])
cdf = scipy.stats.norm.cdf(bins_centers)
pdf = scipy.stats.norm.pdf(bins_centers)
plt.rcParams["font.size"] = 18
fig, ax = plt.subplots(3,1,figsize=(10,16))
ax[0].set_title("cdf")
ax[0].plot(x,y,label="data")
ax[0].plot(x2,y2,label="linear")
ax[0].plot(x3,y3,label="cubic")
ax[0].plot(bins_centers,cdf,label="ans")
ax[1].set_title("pdf:linear")
ax[1].plot(x2b,y2b,label="linear")
ax[1].plot(bins_centers,pdf,label="ans")
ax[2].set_title("pdf:cubic")
ax[2].plot(x3b,y3b,label="cubic")
ax[2].plot(bins_centers,pdf,label="ans")
for idx in range(3):
ax[idx].legend()
setGridLine(ax[idx])
plt.show()
plt.clf()
plt.close()
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2018-04-22 04:36:59
Właśnie do tego wróciłem i musiałem zainstalować scipy jako matplotlib.mlab dał mi komunikat o błędzieMatplotlibDeprecationWarning: scipy.stats.norm.pdf przy próbie przykład powyżej. Więc próbka jest teraz:
%matplotlib inline
import math
import matplotlib.pyplot as plt
import numpy as np
import scipy.stats
mu = 0
variance = 1
sigma = math.sqrt(variance)
x = np.linspace(mu - 3*sigma, mu + 3*sigma, 100)
plt.plot(x, scipy.stats.norm.pdf(x, mu, sigma))
plt.show()
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2018-07-05 09:58:37
Użyj seaborn zamiast używam distplot of seaborn ze średnią=5 std=3 z 1000 wartości
value = np.random.normal(loc=5,scale=3,size=1000)
sns.distplot(value)
Otrzymasz krzywą rozkładu normalnego
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2018-08-09 03:43:51