Importowanie wielu.pliki csv do R
Załóżmy, że mamy folder zawierający wiele danych.pliki csv, każdy zawierający tę samą liczbę zmiennych, ale każdy z różnych czasów. Czy istnieje sposób w R, aby importować je wszystkie jednocześnie, zamiast importować je wszystkie pojedynczo?
Mój problem polega na tym, że mam około 2000 plików danych do zaimportowania i muszę importować je pojedynczo za pomocą kodu:
read.delim(file="filename", header=TRUE, sep="\t")
13 answers
Coś takiego powinno skutkować każdą ramką danych jako oddzielnym elementem na jednej liście:
temp = list.files(pattern="*.csv")
myfiles = lapply(temp, read.delim)
Zakłada to, że masz te pliki CSV w jednym katalogu -- bieżącym katalogu roboczym -- i że wszystkie z nich mają małe litery rozszerzenia .csv.
Jeśli chcesz połączyć te ramki danych w jedną ramkę danych, zobacz rozwiązania w innych odpowiedziach za pomocą takich rzeczy, jak do.call(rbind,...), dplyr::bind_rows() lub data.table::rbindlist().
Jeśli naprawdę chcesz, aby każda ramka danych w oddzielny obiekt, mimo że często jest to niewskazane, możesz wykonać następujące czynności za pomocą assign:
temp = list.files(pattern="*.csv")
for (i in 1:length(temp)) assign(temp[i], read.csv(temp[i]))
Lub, bez assign, i aby zademonstrować (1) Jak można wyczyścić nazwę pliku i (2) Jak używać list2env, Możesz spróbować:
temp = list.files(pattern="*.csv")
list2env(
lapply(setNames(temp, make.names(gsub("*.csv$", "", temp))),
read.csv), envir = .GlobalEnv)
Ale często lepiej zostawić je na jednej liście.
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2018-08-03 19:32:41
Oto kolejna opcja do konwersji .pliki csv w jedno dane.rama. Korzystanie z funkcji bazowych R. Jest to o rząd wielkości wolniejsze niż poniższe opcje.
# Get the files names
files = list.files(pattern="*.csv")
# First apply read.csv, then rbind
myfiles = do.call(rbind, lapply(files, function(x) read.csv(x, stringsAsFactors = FALSE)))
Edit: - kilka dodatkowych opcji za pomocą data.table i readr
A fread() wersja, która jest funkcją pakietu data.table. To powinna być najszybsza opcja.
library(data.table)
DT = do.call(rbind, lapply(files, fread)
# the same using `rbindlist()`
DT = rbindlist(lapply(files, fread))
Używanie readr , czyli nowego pakietu hadley do odczytu plików csv. Trochę wolniejszy niż fread ale z innym funkcjonalności.
library(readr)
library(dplyr)
tbl = lapply(files, read_csv) %>% bind_rows()
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2016-06-18 16:05:25
Szybkie i zwięzłe tidyverse rozwiązanie:
(ponad dwa razy szybciej niż Baza R ' S read.csv)
tbl <-
list.files(pattern = "*.csv") %>%
map_df(~read_csv(.))
data.tabela ' s fread() może nawet skrócić czas ładowania o połowę.
library(data.table)
tbl_fread <-
list.files(pattern = "*.csv") %>%
map_df(~fread(., stringsAsFactors = FALSE))
The stringsAsFactors = FALSE argument utrzymuje współczynnik ramki danych wolny.
Jeśli typecasting jest bezczelny, możesz wymusić, aby wszystkie kolumny były znakami z argumentem col_types.
tbl <-
list.files(pattern = "*.csv") %>%
map_df(~read_csv(., col_types = cols(.default = "c")))
Jeśli chcesz zanurzyć się w podkatalogach, aby skonstruować swój Lista plików, które mają zostać ostatecznie powiązane, a następnie pamiętaj, aby dołączyć nazwę ścieżki, a także zarejestrować pliki z ich pełnymi nazwami na liście. Pozwoli to na kontynuowanie pracy wiązania poza bieżącym katalogiem. (Myślenie o pełnych nazwach ścieżek jako działających jak paszporty, aby umożliwić przemieszczanie się z powrotem przez "granice" katalogu.)
tbl <-
list.files(path = "./subdirectory/",
pattern = "*.csv",
full.names = T) %>%
map_df(~read_csv(., col_types = cols(.default = "c")))
Jak opisuje Hadley tutaj (mniej więcej w połowie):
map_df(x, f)jest skutecznie taki sam jakdo.call("rbind", lapply(x, f))....
Bonus Feature - dodawanie nazw plików do rekordów na żądanie funkcji Niks w komentarzach poniżej:
* Dodaj oryginalny filename do każdego rekordu.
Kod wyjaśniony: Utwórz funkcję do dołączania nazwy pliku do każdego rekordu podczas wstępnego czytania tabel. Następnie użyj tej funkcji zamiast prostej read_csv().
read_plus <- function(flnm) {
read_csv(flnm) %>%
mutate(filename = flnm)
}
tbl_with_sources <-
list.files(pattern = "*.csv",
full.names = T) %>%
map_df(~read_plus(.))
(metody obsługi typecastingu i podkatalogów mogą być również obsługiwane wewnątrz read_plus() funkcja w taki sam sposób, jak pokazano w drugim i trzecim wariancie zaproponowanym powyżej.)
### Benchmark Code & Results
library(tidyverse)
library(data.table)
library(microbenchmark)
### Base R Approaches
#### Instead of a dataframe, this approach creates a list of lists
#### removed from analysis as this alone doubled analysis time reqd
# lapply_read.delim <- function(path, pattern = "*.csv") {
# temp = list.files(path, pattern, full.names = TRUE)
# myfiles = lapply(temp, read.delim)
# }
#### `read.csv()`
do.call_rbind_read.csv <- function(path, pattern = "*.csv") {
files = list.files(path, pattern, full.names = TRUE)
do.call(rbind, lapply(files, function(x) read.csv(x, stringsAsFactors = FALSE)))
}
map_df_read.csv <- function(path, pattern = "*.csv") {
list.files(path, pattern, full.names = TRUE) %>%
map_df(~read.csv(., stringsAsFactors = FALSE))
}
### *dplyr()*
#### `read_csv()`
lapply_read_csv_bind_rows <- function(path, pattern = "*.csv") {
files = list.files(path, pattern, full.names = TRUE)
lapply(files, read_csv) %>% bind_rows()
}
map_df_read_csv <- function(path, pattern = "*.csv") {
list.files(path, pattern, full.names = TRUE) %>%
map_df(~read_csv(., col_types = cols(.default = "c")))
}
### *data.table* / *purrr* hybrid
map_df_fread <- function(path, pattern = "*.csv") {
list.files(path, pattern, full.names = TRUE) %>%
map_df(~fread(., stringsAsFactors = FALSE))
}
### *data.table*
rbindlist_fread <- function(path, pattern = "*.csv") {
files = list.files(path, pattern, full.names = TRUE)
rbindlist(lapply(files, function(x) fread(x, stringsAsFactors = FALSE)))
}
do.call_rbind_fread <- function(path, pattern = "*.csv") {
files = list.files(path, pattern, full.names = TRUE)
do.call(rbind, lapply(files, function(x) fread(x, stringsAsFactors = FALSE)))
}
read_results <- function(dir_size){
microbenchmark(
# lapply_read.delim = lapply_read.delim(dir_size), # too slow to include in benchmarks
do.call_rbind_read.csv = do.call_rbind_read.csv(dir_size),
map_df_read.csv = map_df_read.csv(dir_size),
lapply_read_csv_bind_rows = lapply_read_csv_bind_rows(dir_size),
map_df_read_csv = map_df_read_csv(dir_size),
rbindlist_fread = rbindlist_fread(dir_size),
do.call_rbind_fread = do.call_rbind_fread(dir_size),
map_df_fread = map_df_fread(dir_size),
times = 10L)
}
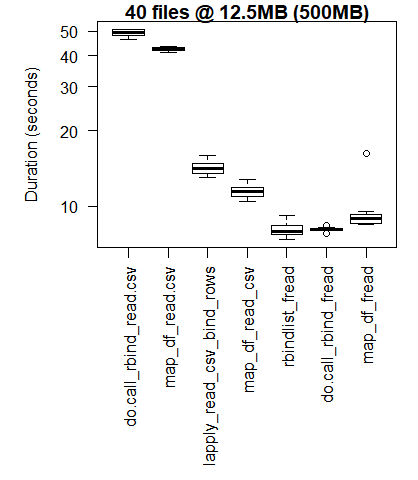
read_results_lrg_mid_mid <- read_results('./testFolder/500MB_12.5MB_40files')
print(read_results_lrg_mid_mid, digits = 3)
read_results_sml_mic_mny <- read_results('./testFolder/5MB_5KB_1000files/')
read_results_sml_tny_mod <- read_results('./testFolder/5MB_50KB_100files/')
read_results_sml_sml_few <- read_results('./testFolder/5MB_500KB_10files/')
read_results_med_sml_mny <- read_results('./testFolder/50MB_5OKB_1000files')
read_results_med_sml_mod <- read_results('./testFolder/50MB_5OOKB_100files')
read_results_med_med_few <- read_results('./testFolder/50MB_5MB_10files')
read_results_lrg_sml_mny <- read_results('./testFolder/500MB_500KB_1000files')
read_results_lrg_med_mod <- read_results('./testFolder/500MB_5MB_100files')
read_results_lrg_lrg_few <- read_results('./testFolder/500MB_50MB_10files')
read_results_xlg_lrg_mod <- read_results('./testFolder/5000MB_50MB_100files')
print(read_results_sml_mic_mny, digits = 3)
print(read_results_sml_tny_mod, digits = 3)
print(read_results_sml_sml_few, digits = 3)
print(read_results_med_sml_mny, digits = 3)
print(read_results_med_sml_mod, digits = 3)
print(read_results_med_med_few, digits = 3)
print(read_results_lrg_sml_mny, digits = 3)
print(read_results_lrg_med_mod, digits = 3)
print(read_results_lrg_lrg_few, digits = 3)
print(read_results_xlg_lrg_mod, digits = 3)
# display boxplot of my typical use case results & basic machine max load
par(oma = c(0,0,0,0)) # remove overall margins if present
par(mfcol = c(1,1)) # remove grid if present
par(mar = c(12,5,1,1) + 0.1) # to display just a single boxplot with its complete labels
boxplot(read_results_lrg_mid_mid, las = 2, xlab = "", ylab = "Duration (seconds)", main = "40 files @ 12.5MB (500MB)")
boxplot(read_results_xlg_lrg_mod, las = 2, xlab = "", ylab = "Duration (seconds)", main = "100 files @ 50MB (5GB)")
# generate 3x3 grid boxplots
par(oma = c(12,1,1,1)) # margins for the whole 3 x 3 grid plot
par(mfcol = c(3,3)) # create grid (filling down each column)
par(mar = c(1,4,2,1)) # margins for the individual plots in 3 x 3 grid
boxplot(read_results_sml_mic_mny, las = 2, xlab = "", ylab = "Duration (seconds)", main = "1000 files @ 5KB (5MB)", xaxt = 'n')
boxplot(read_results_sml_tny_mod, las = 2, xlab = "", ylab = "Duration (milliseconds)", main = "100 files @ 50KB (5MB)", xaxt = 'n')
boxplot(read_results_sml_sml_few, las = 2, xlab = "", ylab = "Duration (milliseconds)", main = "10 files @ 500KB (5MB)",)
boxplot(read_results_med_sml_mny, las = 2, xlab = "", ylab = "Duration (microseconds) ", main = "1000 files @ 50KB (50MB)", xaxt = 'n')
boxplot(read_results_med_sml_mod, las = 2, xlab = "", ylab = "Duration (microseconds)", main = "100 files @ 500KB (50MB)", xaxt = 'n')
boxplot(read_results_med_med_few, las = 2, xlab = "", ylab = "Duration (seconds)", main = "10 files @ 5MB (50MB)")
boxplot(read_results_lrg_sml_mny, las = 2, xlab = "", ylab = "Duration (seconds)", main = "1000 files @ 500KB (500MB)", xaxt = 'n')
boxplot(read_results_lrg_med_mod, las = 2, xlab = "", ylab = "Duration (seconds)", main = "100 files @ 5MB (500MB)", xaxt = 'n')
boxplot(read_results_lrg_lrg_few, las = 2, xlab = "", ylab = "Duration (seconds)", main = "10 files @ 50MB (500MB)")
Middling Use Case
Większy Przypadek Użycia
Różnorodność przypadków użycia
Rows: file counts (1000, 100, 10)
Kolumny: ostateczny rozmiar ramki danych (5MB, 50MB, 500MB)
(kliknij na obrazek, aby zobaczyć oryginalny rozmiar)

Baza R wyniki są lepsze dla najmniejszych przypadki użycia, w których obciążenie bibliotek C purrr i dplyr przeważa nad wzrostem wydajności obserwowanym podczas wykonywania zadań przetwarzania na większą skalę.
Jeśli chcesz uruchomić własne testy, ten skrypt bash może okazać się pomocny.
for ((i=1; i<=$2; i++)); do
cp "$1" "${1:0:8}_${i}.csv";
done
bash what_you_name_this_script.sh "fileName_you_want_copied" 100 utworzy 100 kopii pliku ponumerowanych kolejno (po pierwszych 8 znakach nazwy pliku i podkreślniku).
Uznanie i uznanie
Ze specjalnymi podziękowaniami do:
- Tyler Rinker i Akrun do demonstracji mikrobenchmark.
- Jake Kaupp za wprowadzenie mnie do
map_df()tutaj . - David McLaughlin za pomocną informację zwrotną na temat poprawy wizualizacji i omówienia / potwierdzenia inwersji wydajności zaobserwowanych w małym pliku, wynikach analizy małych ramek danych.
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2018-04-26 22:28:38
Jak również za pomocą lapply lub innego looping construct w R możesz połączyć swoje pliki CSV w jeden plik.
W Unixie, jeśli pliki nie miały nagłówków, to jest tak proste jak:
cat *.csv > all.csv
Lub jeśli są nagłówki i możesz znaleźć ciąg pasujący do nagłówków i tylko nagłówki (tj. przypuśćmy, że wszystkie linie nagłówków zaczynają się od "wieku"), wykonasz:
cat *.csv | grep -v ^Age > all.csv
Myślę, że w Windows można to zrobić z COPY i SEARCH (lub FIND lub coś) z pola poleceń DOS, ale dlaczego nie zainstalować cygwin i uzyskać moc powłoki poleceń Uniksa?
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2012-07-11 13:28:30
Jest to kod, który opracowałem, aby odczytać wszystkie pliki csv do R. utworzy ramkę danych dla każdego pliku csv indywidualnie i tytuł, który ramka danych oryginalnej nazwy pliku (usuwanie spacji i .csv) mam nadzieję, że okaże się to przydatne!
path <- "C:/Users/cfees/My Box Files/Fitness/"
files <- list.files(path=path, pattern="*.csv")
for(file in files)
{
perpos <- which(strsplit(file, "")[[1]]==".")
assign(
gsub(" ","",substr(file, 1, perpos-1)),
read.csv(paste(path,file,sep="")))
}
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2014-02-05 21:44:09
Użycie plyr::ldply powoduje około 50% zwiększenie prędkości poprzez włączenie opcji .parallel Podczas odczytu 400 plików csv o około 30-40 MB każdy. Przykład zawiera pasek postępu tekstu.
library(plyr)
library(data.table)
library(doSNOW)
csv.list <- list.files(path="t:/data", pattern=".csv$", full.names=TRUE)
cl <- makeCluster(4)
registerDoSNOW(cl)
pb <- txtProgressBar(max=length(csv.list), style=3)
pbu <- function(i) setTxtProgressBar(pb, i)
dt <- setDT(ldply(csv.list, fread, .parallel=TRUE, .paropts=list(.options.snow=list(progress=pbu))))
stopCluster(cl)
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2016-12-16 23:55:51
Moim zdaniem większość innych odpowiedzi jest przestarzała przez rio::import_list, która jest zwięzłą jednowierszową:
library(rio)
my_data <- import_list(dir("path_to_directory", pattern = ".csv", rbind = TRUE))
Wszelkie dodatkowe argumenty są przekazywane do rio::import. {[3] } może poradzić sobie z prawie każdym formatem pliku, który R może odczytać, i używa data.table'S fread, gdzie to możliwe, więc powinien być również szybki.
Bazując na komentarzu dnlbrk, przypisywanie może być znacznie szybsze niż list2env dla dużych plików.
library(readr)
library(stringr)
List_of_file_paths <- list.files(path ="C:/Users/Anon/Documents/Folder_with_csv_files/", pattern = ".csv", all.files = TRUE, full.names = TRUE)
Ustawiając pełną.nazwa argumentu do true, otrzymasz pełną ścieżkę do każdego pliku jako osobny ciąg znaków na liście plików, np. List_of_file_paths[1] będzie coś w stylu "C:/Users/Anon/Documents/Folder_with_csv_files/file1.csv "
for(f in 1:length(List_of_filepaths)) {
file_name <- str_sub(string = List_of_filepaths[f], start = 46, end = -5)
file_df <- read_csv(List_of_filepaths[f])
assign( x = file_name, value = file_df, envir = .GlobalEnv)
}
for(f in 1:length(List_of_filepaths)) {
file_name <- str_sub(string = List_of_filepaths[f], start = 46, end = -5)
file_df <- read_csv(List_of_filepaths[f])
file_df <- file_df[,1:3] #if you only need the first three columns
assign( x = file_name, value = file_df, envir = .GlobalEnv)
}
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2016-09-24 10:23:08
Mój fork zaakceptowanej odpowiedzi jest nieco szybszy i usuwa .csv z nazwy obiektu w R.
temp = list.files(pattern="*.csv")
for (i in 1:length(temp)) assign(gsub(".csv", "", temp[i]), read_csv(temp[i]))
Opiera się na pakiecie readr (tj. library(readr)). Możesz zrobić coś podobnego z data.table Jeśli chcesz.
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2018-05-27 14:28:53
Używam tego z powodzeniem:
xlist<-list.files(pattern = "*.csv")
for(i in xlist) {
x <- read.csv((i))
assign(i, x)
}
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2016-05-06 07:54:39
Jeśli chcesz zebrać różne pliki csv w jedno dane.ramka, można użyć następujących. zauważ, że dane "x".ramka powinna być utworzona z wyprzedzeniem.
temp <- list.files(pattern="*.csv")
for (i in 1:length(temp)) {
temp2 = read.csv(temp[i], header = TRUE)
x <- rbind(x,temp2)
}
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2016-05-19 06:14:05
Możesz użyć do tego pakietu superb sparklyr:
# RStudio will help you get set-up with the Spark dependencies
library(sparklyr)
library(dplyr)
sc <- spark_connect(master = "local", version = "2.0.2")
df <- spark_read_csv(sc,
"dummy",
"file:////Users/bob/dev/data/results/*/*/*-metrics.csv") %>%
collect()
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2017-01-27 12:21:33
To część mojego scenariusza.
#This cycle read the files in a directory and assign the filenames to datasets
files <- list.files(pattern=".csv$")
for(i in files) {
X <- read.table(i, header=TRUE)
SN<-X$A/X$B
X<-cbind(X,SN)
ds<-paste("data_",i, sep="")#this add "data_" to the name of file
ds<-substr(ds, 1, nchar(ds)-4)#remove the last 4 char (.csv)
assign(ds, X)
}
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2012-07-11 14:18:33