Wyhodowanie danych.kadrowanie w sposób efektywny pamięciowo
Zgodnie z tworzeniem ramki danych r wiersz po wierszu , nie jest idealnym rozwiązaniem dołączanie do data.frame za pomocą rbind, ponieważ tworzy ona kopię całych danych.kadruj za każdym razem. Jak gromadzić dane w R w wyniku data.frame bez ponoszenia tej kary? Format pośredni nie musi być data.frame.
4 answers
Pierwsze podejście
Próbowałem uzyskać dostęp do każdego elementu wstępnie przydzielonych danych.frame:
res <- data.frame(x=rep(NA,1000), y=rep(NA,1000))
tracemem(res)
for(i in 1:1000) {
res[i,"x"] <- runif(1)
res[i,"y"] <- rnorm(1)
}
Ale tracemem szaleje (np. dane.ramka jest kopiowana na nowy adres za każdym razem).
Alternatywne podejście (również nie działa)
Jednym z podejść (nie jestem pewien, czy jest szybszy, ponieważ nie mam jeszcze benchmarked) jest stworzenie listy danych.ramki, a następnie stack wszystkie razem:
makeRow <- function() data.frame(x=runif(1),y=rnorm(1))
res <- replicate(1000, makeRow(), simplify=FALSE ) # returns a list of data.frames
library(taRifx)
res.df <- stack(res)
Niestety tworząc listę myślę, że będzie ciężko przeznaczyć na wstępną alokację. Na przykład:
> tracemem(res)
[1] "<0x79b98b0>"
> res[[2]] <- data.frame()
tracemem[0x79b98b0 -> 0x71da500]:
Innymi słowy, zastąpienie elementu listy powoduje skopiowanie listy. Zakładam, że cała lista, ale możliwe, że to tylko ten element listy. Nie znam szczegółów zarządzania pamięcią R.
Prawdopodobnie najlepsze podejście
Jak w przypadku wielu procesów o ograniczonej szybkości lub pamięci, najlepszym rozwiązaniem może być użycie data.table zamiast data.frame. Od data.table ma := przypisać przez operatora odniesienia, może aktualizować bez ponownego kopiowania:
library(data.table)
dt <- data.table(x=rep(0,1000), y=rep(0,1000))
tracemem(dt)
for(i in 1:1000) {
dt[i,x := runif(1)]
dt[i,y := rnorm(1)]
}
# note no message from tracemem
Ale jak wskazuje @ MatthewDowle, set() jest właściwym sposobem, aby to zrobić wewnątrz pętli. To sprawia, że jeszcze szybciej:
library(data.table)
n <- 10^6
dt <- data.table(x=rep(0,n), y=rep(0,n))
dt.colon <- function(dt) {
for(i in 1:n) {
dt[i,x := runif(1)]
dt[i,y := rnorm(1)]
}
}
dt.set <- function(dt) {
for(i in 1:n) {
set(dt,i,1L, runif(1) )
set(dt,i,2L, rnorm(1) )
}
}
library(microbenchmark)
m <- microbenchmark(dt.colon(dt), dt.set(dt),times=2)
(wyniki pokazane poniżej)
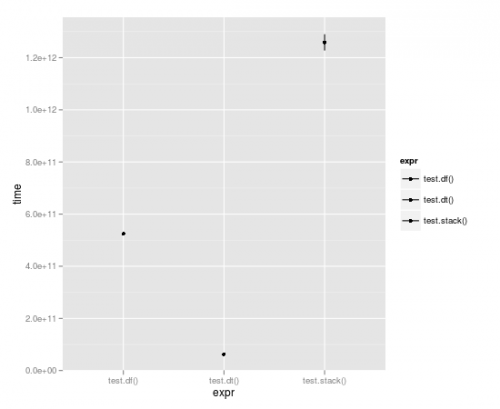
Benchmarking
Z pętlą wykonaną 10 000 razy, Tabela danych jest prawie o pełny rząd wielkości szybsza:Unit: seconds
expr min lq median uq max
1 test.df() 523.49057 523.49057 524.52408 525.55759 525.55759
2 test.dt() 62.06398 62.06398 62.98622 63.90845 63.90845
3 test.stack() 1196.30135 1196.30135 1258.79879 1321.29622 1321.29622
I porównanie := z set():
> m
Unit: milliseconds
expr min lq median uq max
1 dt.colon(dt) 654.54996 654.54996 656.43429 658.3186 658.3186
2 dt.set(dt) 13.29612 13.29612 15.02891 16.7617 16.7617
Zauważ, że n tutaj jest 10^6 a nie 10^5 jak w benchmarkach wykreślonych powyżej. Więc jest rząd wielkości więcej pracy, a wynik jest mierzony w milisekundach, a nie sekundach. Imponujące.
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2012-07-18 11:04:28
Możesz również mieć obiekt pustej listy, w którym elementy są wypełnione ramkami danych; następnie zbierz wyniki na końcu za pomocą sapply lub podobnych. Przykład można znaleźć tutaj. Nie pociągnie to za sobą kar za uprawianie przedmiotu.
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2012-07-14 20:09:02
Jestem bardzo zaskoczony, że nikt jeszcze nie wspomniał o konwersji do macierzy...
W porównaniu z dt.dwukropek i dt.zbiór funkcji zdefiniowanych przez Ariego B. Friedmana , konwersja na macierz ma najlepszy czas działania (nieco szybszy niż dt.dwukropek ). Wszystkie afektacje wewnątrz macierzy są wykonywane przez odniesienie, Więc nie ma niepotrzebnej kopii pamięci wykonywanej w tym kodzie.
Kod:
library(data.table)
n <- 10^4
dt <- data.table(x=rep(0,n), y=rep(0,n))
use.matrix <- function(dt) {
mat = as.matrix(dt) # converting to matrix
for(i in 1:n) {
mat[i,1] = runif(1)
mat[i,2] = rnorm(1)
}
return(as.data.frame(mat)) # converting back to a data.frame
}
dt.colon <- function(dt) { # same as Ari's function
for(i in 1:n) {
dt[i,x := runif(1)]
dt[i,y := rnorm(1)]
}
}
dt.set <- function(dt) { # same as Ari's function
for(i in 1:n) {
set(dt,i,1L, runif(1) )
set(dt,i,2L, rnorm(1) )
}
}
library(microbenchmark)
microbenchmark(dt.colon(dt), dt.set(dt), use.matrix(dt),times=10)
Wynik:
Unit: milliseconds
expr min lq median uq max neval
dt.colon(dt) 7107.68494 7193.54792 7262.76720 7277.24841 7472.41726 10
dt.set(dt) 93.25954 94.10291 95.07181 97.09725 99.18583 10
use.matrix(dt) 48.15595 51.71100 52.39375 54.59252 55.04192 10
Plusy użycie macierzy:
- jest to najszybsza Metoda do tej pory
- nie musisz uczyć się/używać danych.obiekty tabeli
Con użycia macierzy:
- można obsługiwać tylko jeden typ danych w macierzy (w szczególności, jeśli w kolumnach danych były mieszane typy.ramki, wtedy wszystkie zostaną przekonwertowane na znak w linii: mat = as.macierz (dt) # konwersja na macierz )
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2017-05-23 12:25:55
Lubię RSQLite w tej kwestii: dbWriteTable(...,append=TRUE) oświadczenia Podczas zbierania, i dbReadTable Oświadczenie na końcu.
Jeśli dane są wystarczająco małe, można użyć pliku": memory:", jeśli jest duży, dysk twardy.
Oczywiście nie może konkurować pod względem prędkości:
makeRow <- function() data.frame(x=runif(1),y=rnorm(1))
library(RSQLite)
con <- dbConnect(RSQLite::SQLite(), ":memory:")
collect1 <- function(n) {
for (i in 1:n) dbWriteTable(con, "test", makeRow(), append=TRUE)
dbReadTable(con, "test", row.names=NULL)
}
collect2 <- function(n) {
res <- data.frame(x=rep(NA, n), y=rep(NA, n))
for(i in 1:n) res[i,] <- makeRow()[1,]
res
}
> system.time(collect1(1000))
User System verstrichen
7.01 0.00 7.05
> system.time(collect2(1000))
User System verstrichen
0.80 0.01 0.81
Ale może lepiej wyglądać, jeśli data.frame s mają więcej niż jeden rząd. I nie musisz znać liczby wierszy z góry.
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2015-01-21 05:06:36