Dobre sposoby wizualizacji podłużnych danych kategorycznych w R
[Update: chociaż zaakceptowałem odpowiedź, proszę dodać inną odpowiedź, jeśli masz dodatkowe pomysły wizualizacji(czy to w R lub innym języku / programie). Teksty na temat analizy danych kategorycznych nie wydają się mówić wiele o wizualizacji danych podłużnych, podczas gdy teksty na temat analizy danych podłużnych nie wydają się mówić wiele o wizualizacji wewnątrz przedmiotu zmian w czasie w członkostwie kategorii. Więcej odpowiedzi na to pytanie sprawi, że będzie to lepszy zasób w kwestii, która nie ma większego zasięgu w standardowych referencjach.]
Kolega właśnie dał mi Podłużny kategoryczny zestaw danych do obejrzenia i próbuję dowiedzieć się, jak uchwycić Podłużny aspekt w wizualizacji. Zamieszczam tutaj, ponieważ chciałbym to zrobić w R, ale proszę dać mi znać, jeśli ma sens również cross-post Do Cross-Validated, ponieważ Cross-post jest ogólnie zniechęcany.
Szybkie tło: dane śledzą pozycję akademicką studentów od semestru do semestru który przeszedł akademicki program doradczy. Dane są w długim formacie i mają pięć zmiennych: "id", "cohort", "term", "standing"i " termGPA". Pierwsze dwa identyfikują ucznia i termin, w którym był w programie doradczym. Ostatnie trzy to terminy, w których notowano pozycję akademicką studenta i średnią ocen. Wkleiłem kilka przykładowych danych poniżej używając dput.
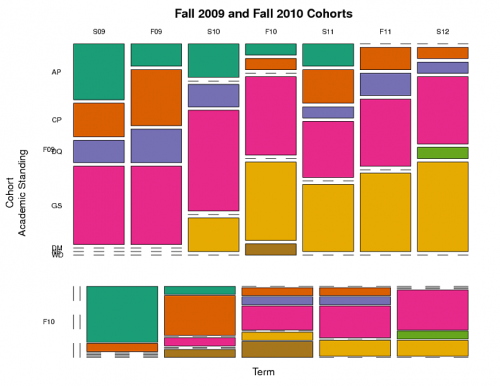
Stworzyłem mozaikę (patrz poniżej), która grupuje uczniów według kohorty, pozycji i terminu. To pokazuje jaka część studentów była w każdej kategorii akademickiej w każdej kadencji. Ale to nie oddaje aspektu podłużnego-fakt, że poszczególni uczniowie są śledzeni w czasie. Chciałbym śledzić drogę, którą grupy studentów o danej pozycji akademickiej podejmują w czasie.
Na przykład: studentów z stałym " AP " (okres próbny) jesienią 2009 ("F09"), jaka frakcja była nadal AP w przyszłości, a jaka frakcja przeniosła się do innych kategorii (np. standing")? Czy istnieją różnice między kohortami w zakresie przemieszczania się między kategoriami w czasie od wejścia do programu doradczego?
Nie mogłem do końca zrozumieć, jak uchwycić ten Podłużny aspekt w grafice R. Pakiet vcd ma możliwości wizualizacji danych kategorycznych, ale wydaje się, że nie adresuje podłużnych danych kategorycznych. Czy istnieją "standardowe" metody wizualizacji podłużnych danych kategorycznych? Czy R ma pakiety przeznaczone do tego celu? Jest długa format odpowiedni dla tego typu danych, czy lepiej byłoby z szerokim formatem?
Byłbym wdzięczny za sugestie dotyczące rozwiązania tego konkretnego problemu, a także sugestie dotyczące artykułów, książek itp. aby dowiedzieć się więcej o wizualizacji podłużnych danych kategorycznych.
Oto kod, którego użyłem do stworzenia mozaiki. Kod wykorzystuje dane wymienione poniżej zdput.
library(RColorBrewer)
# create a table object for plotting
df1.tab = table(df1$cohort, df1$term, df1$standing,
dnn=c("Cohort\nAcademic Standing", "Term", "Standing"))
# create a mosaic plot
plot(df1.tab, las=1, dir=c("h","v","h"),
col=brewer.pal(8,"Dark2"),
main="Fall 2009 and Fall 2010 Cohorts")
Oto wykres mozaiki (pytanie poboczne: czy jest jakiś sposób na wykonanie kolumn dla kohorty F10 siedzieć bezpośrednio pod i mieć taką samą szerokość jak kolumny dla kohorty F09, nawet jeśli nie ma danych dla niektórych terminów w kohorcie F10?):
A oto dane użyte do utworzenia tabeli i wykresu:
df1 =
structure(list(id = c(101L, 102L, 103L, 104L, 105L, 106L, 107L,
108L, 109L, 110L, 111L, 112L, 113L, 114L, 115L, 116L, 117L, 118L,
119L, 120L, 121L, 122L, 123L, 124L, 125L, 101L, 102L, 103L, 104L,
105L, 106L, 107L, 108L, 109L, 110L, 111L, 112L, 113L, 114L, 115L,
116L, 117L, 118L, 119L, 120L, 121L, 122L, 123L, 124L, 125L, 101L,
102L, 103L, 104L, 105L, 106L, 107L, 108L, 109L, 110L, 111L, 112L,
113L, 114L, 115L, 116L, 117L, 118L, 119L, 120L, 121L, 122L, 123L,
124L, 125L, 101L, 102L, 103L, 104L, 105L, 106L, 107L, 108L, 109L,
110L, 111L, 112L, 113L, 114L, 115L, 116L, 117L, 118L, 119L, 120L,
121L, 122L, 123L, 124L, 125L, 101L, 102L, 103L, 104L, 105L, 106L,
107L, 108L, 109L, 110L, 111L, 112L, 113L, 114L, 115L, 116L, 117L,
118L, 119L, 120L, 121L, 122L, 123L, 124L, 125L, 101L, 102L, 103L,
104L, 105L, 106L, 107L, 108L, 109L, 110L, 111L, 112L, 113L, 114L,
115L, 116L, 117L, 118L, 119L, 120L, 121L, 122L, 123L, 124L, 125L,
101L, 102L, 103L, 104L, 105L, 106L, 107L, 108L, 109L, 110L, 111L,
112L, 113L, 114L, 115L, 116L, 117L, 118L, 119L, 120L, 121L, 122L,
123L, 124L, 125L), cohort = structure(c(1L, 1L, 1L, 1L, 2L, 1L,
1L, 2L, 2L, 2L, 2L, 1L, 1L, 1L, 1L, 1L, 1L, 1L, 2L, 2L, 1L, 1L,
1L, 1L, 2L, 1L, 1L, 1L, 1L, 2L, 1L, 1L, 2L, 2L, 2L, 2L, 1L, 1L,
1L, 1L, 1L, 1L, 1L, 2L, 2L, 1L, 1L, 1L, 1L, 2L, 1L, 1L, 1L, 1L,
2L, 1L, 1L, 2L, 2L, 2L, 2L, 1L, 1L, 1L, 1L, 1L, 1L, 1L, 2L, 2L,
1L, 1L, 1L, 1L, 2L, 1L, 1L, 1L, 1L, 2L, 1L, 1L, 2L, 2L, 2L, 2L,
1L, 1L, 1L, 1L, 1L, 1L, 1L, 2L, 2L, 1L, 1L, 1L, 1L, 2L, 1L, 1L,
1L, 1L, 2L, 1L, 1L, 2L, 2L, 2L, 2L, 1L, 1L, 1L, 1L, 1L, 1L, 1L,
2L, 2L, 1L, 1L, 1L, 1L, 2L, 1L, 1L, 1L, 1L, 2L, 1L, 1L, 2L, 2L,
2L, 2L, 1L, 1L, 1L, 1L, 1L, 1L, 1L, 2L, 2L, 1L, 1L, 1L, 1L, 2L,
1L, 1L, 1L, 1L, 2L, 1L, 1L, 2L, 2L, 2L, 2L, 1L, 1L, 1L, 1L, 1L,
1L, 1L, 2L, 2L, 1L, 1L, 1L, 1L, 2L), .Label = c("F09", "F10"), class = c("ordered",
"factor")), term = structure(c(1L, 1L, 1L, 1L, 1L, 1L, 1L, 1L,
1L, 1L, 1L, 1L, 1L, 1L, 1L, 1L, 1L, 1L, 1L, 1L, 1L, 1L, 1L, 1L,
1L, 2L, 2L, 2L, 2L, 2L, 2L, 2L, 2L, 2L, 2L, 2L, 2L, 2L, 2L, 2L,
2L, 2L, 2L, 2L, 2L, 2L, 2L, 2L, 2L, 2L, 3L, 3L, 3L, 3L, 3L, 3L,
3L, 3L, 3L, 3L, 3L, 3L, 3L, 3L, 3L, 3L, 3L, 3L, 3L, 3L, 3L, 3L,
3L, 3L, 3L, 4L, 4L, 4L, 4L, 4L, 4L, 4L, 4L, 4L, 4L, 4L, 4L, 4L,
4L, 4L, 4L, 4L, 4L, 4L, 4L, 4L, 4L, 4L, 4L, 4L, 5L, 5L, 5L, 5L,
5L, 5L, 5L, 5L, 5L, 5L, 5L, 5L, 5L, 5L, 5L, 5L, 5L, 5L, 5L, 5L,
5L, 5L, 5L, 5L, 5L, 6L, 6L, 6L, 6L, 6L, 6L, 6L, 6L, 6L, 6L, 6L,
6L, 6L, 6L, 6L, 6L, 6L, 6L, 6L, 6L, 6L, 6L, 6L, 6L, 6L, 7L, 7L,
7L, 7L, 7L, 7L, 7L, 7L, 7L, 7L, 7L, 7L, 7L, 7L, 7L, 7L, 7L, 7L,
7L, 7L, 7L, 7L, 7L, 7L, 7L), .Label = c("S09", "F09", "S10",
"F10", "S11", "F11", "S12"), class = c("ordered", "factor")),
standing = structure(c(2L, 4L, 1L, 4L, NA, 4L, 1L, NA, NA,
NA, NA, 2L, 2L, 1L, 4L, 4L, 1L, 3L, NA, NA, 4L, 3L, 1L, 4L,
NA, 2L, 1L, 3L, 3L, NA, 1L, 2L, NA, NA, NA, NA, 2L, 4L, 3L,
4L, 4L, 4L, 2L, NA, NA, 4L, 2L, 4L, 4L, NA, 3L, 4L, 6L, 6L,
1L, 4L, 4L, 1L, 1L, 1L, 1L, 1L, 4L, 6L, 4L, 4L, 1L, 4L, 1L,
2L, 4L, 3L, 1L, 4L, 1L, 6L, 1L, 6L, 6L, 7L, 4L, 4L, 2L, 2L,
4L, 2L, 6L, 4L, 6L, 7L, 4L, 2L, 4L, 1L, 2L, 4L, 6L, 6L, 4L,
2L, 2L, 3L, 6L, 6L, 7L, 4L, 4L, 3L, 4L, 4L, 6L, 2L, 1L, 6L,
6L, 4L, 2L, 1L, 7L, 2L, 4L, 6L, 6L, 4L, 4L, 3L, 6L, 4L, 6L,
2L, 4L, 4L, 6L, 4L, 4L, 6L, 3L, 2L, 6L, 6L, 4L, 2L, 6L, 3L,
4L, 4L, 6L, 6L, 4L, 4L, 5L, 6L, 4L, 6L, 4L, 4L, 4L, 5L, 4L,
4L, 6L, 6L, 2L, 6L, 6L, 4L, 3L, 6L, 6L, 4L, 4L, 6L, 6L, 4L,
4L), .Label = c("AP", "CP", "DQ", "GS", "DM", "NE", "WD"), class = "factor"),
termGPA = c(1.433, 1.925, 1, 1.68, NA, 1.579, 1.233, NA,
NA, NA, NA, 2.009, 1.675, 0, 1.5, 1.86, 0.5, 0.94, NA, NA,
1.777, 1.1, 1.133, 1.675, NA, 2, 1.25, 1.66, 0, NA, 1.525,
2.25, NA, NA, NA, NA, 1.66, 2.325, 0, 2.308, 1.6, 1.825,
2.33, NA, NA, 2.65, 2.65, 2.85, 3.233, NA, 1.25, 1.575, NA,
NA, 1, 2.385, 3.133, 0, 0, 1.729, 1.075, 0, 4, NA, 2.74,
0, 1.369, 2.53, 0, 2.65, 2.75, 0, 0.333, 3.367, 1, NA, 0.1,
NA, NA, 1, 2.2, 2.18, 2.31, 1.75, 3.073, 0.7, NA, 1.425,
NA, 2.74, 2.9, 0.692, 2, 0.75, 1.675, 2.4, NA, NA, 3.829,
2.33, 2.3, 1.5, NA, NA, NA, 2.69, 1.52, 0.838, 2.35, 1.55,
NA, 1.35, 0.66, NA, NA, 1.35, 1.9, 1.04, NA, 1.464, 2.94,
NA, NA, 3.72, 2.867, 1.467, NA, 3.133, NA, 1, 2.458, 1.214,
NA, 3.325, 2.315, NA, 1, 2.233, NA, NA, 2.567, 1, NA, 0,
3.325, 2.077, NA, NA, 3.85, 2.718, 1.385, NA, 2.333, NA,
2.675, 1.267, 1.6, 1.388, 3.433, 0.838, NA, NA, 0, NA, NA,
2.6, 0, NA, NA, 1, 2.825, NA, NA, 3.838, 2.883)), .Names = c("id",
"cohort", "term", "standing", "termGPA"), row.names = c("101.F09.s09",
"102.F09.s09", "103.F09.s09", "104.F09.s09", "105.F10.s09", "106.F09.s09",
"107.F09.s09", "108.F10.s09", "109.F10.s09", "110.F10.s09", "111.F10.s09",
"112.F09.s09", "113.F09.s09", "114.F09.s09", "115.F09.s09", "116.F09.s09",
"117.F09.s09", "118.F09.s09", "119.F10.s09", "120.F10.s09", "121.F09.s09",
"122.F09.s09", "123.F09.s09", "124.F09.s09", "125.F10.s09", "101.F09.f09",
"102.F09.f09", "103.F09.f09", "104.F09.f09", "105.F10.f09", "106.F09.f09",
"107.F09.f09", "108.F10.f09", "109.F10.f09", "110.F10.f09", "111.F10.f09",
"112.F09.f09", "113.F09.f09", "114.F09.f09", "115.F09.f09", "116.F09.f09",
"117.F09.f09", "118.F09.f09", "119.F10.f09", "120.F10.f09", "121.F09.f09",
"122.F09.f09", "123.F09.f09", "124.F09.f09", "125.F10.f09", "101.F09.s10",
"102.F09.s10", "103.F09.s10", "104.F09.s10", "105.F10.s10", "106.F09.s10",
"107.F09.s10", "108.F10.s10", "109.F10.s10", "110.F10.s10", "111.F10.s10",
"112.F09.s10", "113.F09.s10", "114.F09.s10", "115.F09.s10", "116.F09.s10",
"117.F09.s10", "118.F09.s10", "119.F10.s10", "120.F10.s10", "121.F09.s10",
"122.F09.s10", "123.F09.s10", "124.F09.s10", "125.F10.s10", "101.F09.f10",
"102.F09.f10", "103.F09.f10", "104.F09.f10", "105.F10.f10", "106.F09.f10",
"107.F09.f10", "108.F10.f10", "109.F10.f10", "110.F10.f10", "111.F10.f10",
"112.F09.f10", "113.F09.f10", "114.F09.f10", "115.F09.f10", "116.F09.f10",
"117.F09.f10", "118.F09.f10", "119.F10.f10", "120.F10.f10", "121.F09.f10",
"122.F09.f10", "123.F09.f10", "124.F09.f10", "125.F10.f10", "101.F09.s11",
"102.F09.s11", "103.F09.s11", "104.F09.s11", "105.F10.s11", "106.F09.s11",
"107.F09.s11", "108.F10.s11", "109.F10.s11", "110.F10.s11", "111.F10.s11",
"112.F09.s11", "113.F09.s11", "114.F09.s11", "115.F09.s11", "116.F09.s11",
"117.F09.s11", "118.F09.s11", "119.F10.s11", "120.F10.s11", "121.F09.s11",
"122.F09.s11", "123.F09.s11", "124.F09.s11", "125.F10.s11", "101.F09.f11",
"102.F09.f11", "103.F09.f11", "104.F09.f11", "105.F10.f11", "106.F09.f11",
"107.F09.f11", "108.F10.f11", "109.F10.f11", "110.F10.f11", "111.F10.f11",
"112.F09.f11", "113.F09.f11", "114.F09.f11", "115.F09.f11", "116.F09.f11",
"117.F09.f11", "118.F09.f11", "119.F10.f11", "120.F10.f11", "121.F09.f11",
"122.F09.f11", "123.F09.f11", "124.F09.f11", "125.F10.f11", "101.F09.s12",
"102.F09.s12", "103.F09.s12", "104.F09.s12", "105.F10.s12", "106.F09.s12",
"107.F09.s12", "108.F10.s12", "109.F10.s12", "110.F10.s12", "111.F10.s12",
"112.F09.s12", "113.F09.s12", "114.F09.s12", "115.F09.s12", "116.F09.s12",
"117.F09.s12", "118.F09.s12", "119.F10.s12", "120.F10.s12", "121.F09.s12",
"122.F09.s12", "123.F09.s12", "124.F09.s12", "125.F10.s12"), reshapeLong = structure(list(
varying = list(c("s09as", "f09as", "s10as", "f10as", "s11as",
"f11as", "s12as"), c("s09termGPA", "f09termGPA", "s10termGPA",
"f10termGPA", "s11termGPA", "f11termGPA", "s12termGPA")),
v.names = c("standing", "termGPA"), idvar = c("id", "cohort"
), timevar = "term"), .Names = c("varying", "v.names", "idvar",
"timevar")), class = "data.frame")
3 answers
Oto kilka pomysłów na wykreśl swoje dane. Użyłem ggplot2 i trochę sformatowałem dane w miejscach.
Rysunek 1
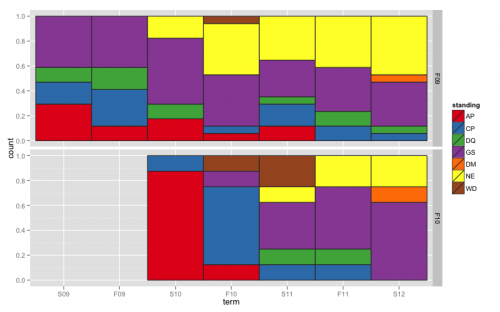 Użyłem ułożonego słupka do naśladowania Twojej mozaiki i rozwiązania problemu wyrównania.
Użyłem ułożonego słupka do naśladowania Twojej mozaiki i rozwiązania problemu wyrównania.
Rysunek 2
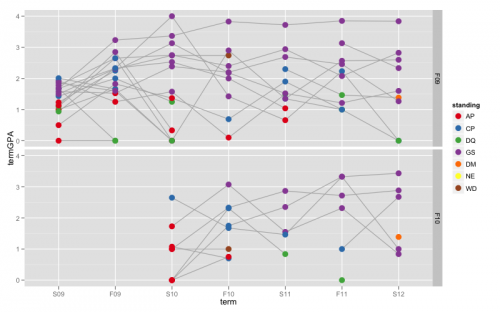 Punkty danych dla każdego ucznia są połączone szarą linią, co przypomina równoległy Wykres współrzędnych. Kolorowanie punktów pokazuje kategoryczną pozycję. Korzystanie z GPA na osi y pomaga rozłożyć punkty aby zmniejszyć nadmiar i pokazuje korelację pozycji stojącej i GPA. Głównym problemem jest to, że wiele ważnych
Punkty danych dla każdego ucznia są połączone szarą linią, co przypomina równoległy Wykres współrzędnych. Kolorowanie punktów pokazuje kategoryczną pozycję. Korzystanie z GPA na osi y pomaga rozłożyć punkty aby zmniejszyć nadmiar i pokazuje korelację pozycji stojącej i GPA. Głównym problemem jest to, że wiele ważnych standing datapointów traci ważność, ponieważ brakuje im pasującej wartości termGPA.
Rysunek 3
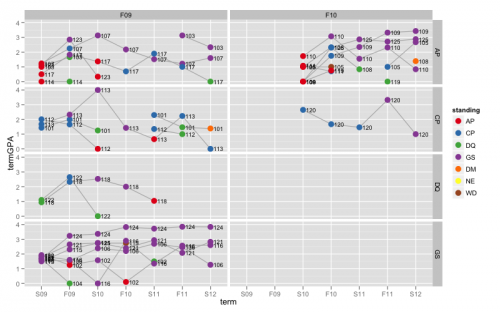 Tutaj utworzyłem nową zmienną o nazwie initial_standing do użycia do facettingu. Każdy panel zawiera uczniów, którzy pasują zarówno do cohort, jak i initial_standing. Wykreśl id jako tekst sprawia, że liczba ta jest nieco zaśmiecona, ale może być przydatna w niektórych przypadkach.
Tutaj utworzyłem nową zmienną o nazwie initial_standing do użycia do facettingu. Każdy panel zawiera uczniów, którzy pasują zarówno do cohort, jak i initial_standing. Wykreśl id jako tekst sprawia, że liczba ta jest nieco zaśmiecona, ale może być przydatna w niektórych przypadkach.
Rysunek 4
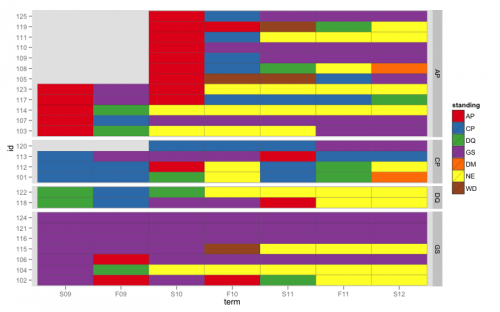 Ten wykres jest jak heatmapa, gdzie każdy wiersz jest studentem. Kontrolowałem kolejność osi
Ten wykres jest jak heatmapa, gdzie każdy wiersz jest studentem. Kontrolowałem kolejność osi id, aby wymusić, aby grupy initial_standing i cohort pozostały razem. Jeśli masz więcej wierszy, możesz rozważyć sortowanie wierszy według pewnego rodzaju klastrów.
library(ggplot2)
# Create new data frame for determining initial standing.
standing_data = data.frame(id=unique(df1$id), initial_standing=NA, cohort=NA)
for (i in 1:nrow(standing_data)) {
id = standing_data$id[i]
subdat = df1[df1$id == id, ]
subdat = subdat[complete.cases(subdat), ]
initial_standing = subdat$standing[which.min(subdat$term)]
standing_data[i, "initial_standing"] = as.character(initial_standing)
standing_data[i, "cohort"] = as.character(subdat$cohort[1])
}
standing_data$cohort = factor(standing_data$cohort, levels=levels(df1$cohort))
standing_data$initial_standing = factor(standing_data$initial_standing,
levels=levels(df1$standing))
# Add the new column (initial_standing) to df1.
df1 = merge(df1, standing_data[, c("id", "initial_standing")], by="id")
# Remove rows where standing is missing. Make some plots tidier.
df1 = df1[!is.na(df1$standing), ]
# Create id factor, controlling the sort order of the levels.
id_order = order(standing_data$initial_standing, standing_data$cohort)
df1$id = factor(df1$id, levels=as.character(standing_data$id)[id_order])
p1 = ggplot(df1, aes(x=term, fill=standing)) +
geom_bar(position="fill", colour="grey20", size=0.5, width=1.0) +
facet_grid(cohort ~ .) +
scale_fill_brewer(palette="Set1")
p2 = ggplot(df1, aes(x=term, y=termGPA, group=id)) +
geom_line(colour="grey70") +
geom_point(aes(colour=standing), size=4) +
facet_grid(cohort ~ .) +
scale_colour_brewer(palette="Set1")
p3 = ggplot(df1, aes(x=term, y=termGPA, group=id)) +
geom_line(colour="grey70") +
geom_point(aes(colour=standing), size=4) +
geom_text(aes(label=id), hjust=-0.30, size=3) +
facet_grid(initial_standing ~ cohort) +
scale_colour_brewer(palette="Set1")
p4 = ggplot(df1, aes(x=term, y=id, fill=standing)) +
geom_tile(colour="grey20") +
facet_grid(initial_standing ~ ., space="free_y", scales="free_y") +
scale_fill_brewer(palette="Set1") +
opts(panel.grid.major=theme_blank()) +
opts(panel.grid.minor=theme_blank())
ggsave("plot_1.png", p1, width=10, height=6.25, dpi=80)
ggsave("plot_2.png", p2, width=10, height=6.25, dpi=80)
ggsave("plot_3.png", p3, width=10, height=6.25, dpi=80)
ggsave("plot_4.png", p4, width=10, height=6.25, dpi=80)
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2012-07-17 07:38:09
Badając moje pytanie, znalazłem kilka innych opcji, które wymienię tutaj.
Wiele stosunkowo nowych pakietów R jest przeznaczonych do wizualizacji i analizy danych "historii życia" lub "sekwencji wielostanowych". Chodzi o to, że z biegiem czasu ludzie (lub przedmioty) wchodzą i wychodzą z różnych kategorii-na przykład zmiany w karierze, małżeństwo i rozwód, zdrowie i choroby lub, w moim przypadku, kategorie akademickiej pozycji w college ' u.
R pakiety do wizualizacji sekwencji lub życia dane historyczne obejmują biograf, wspomniany przez @timriffe w komentarzu powyżej, i TraMineR. Autor pakietu biograph, Frans Willekens, ma książkę na opakowaniu, Biograph. Wielostanowa analiza historii życia z R , która zostanie opublikowana przez Springer tej jesieni. TraMineR ma szczegółową instrukcję obsługi pod linkiem powyżej, a także krótszy artykuł JSS . JSS ma również specjalne wydanie dotyczące modeli wielostanowiskowych w kontekście analizy ryzyka , które omawia dodatkowe pakiety R do modelowania wielostanowego.
Znalazłem również wyspecjalizowane oprogramowanie zaprojektowane do wizualizacji ruchów między kategoriami w czasie. Parallel Sets to prosty, darmowy program do tworzenia podstawowych wizualizacji, choć ma ograniczoną elastyczność. Lifeflow jest bardziej wyrafinowany. Jest również bezpłatny, ale musisz wysłać e-mail do twórcy z prośbą o kopię.
Dodam więcej szczegółów do tej odpowiedzi, gdy tylko będę miał okazję wypróbować te narzędzia.
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2012-07-18 18:12:19
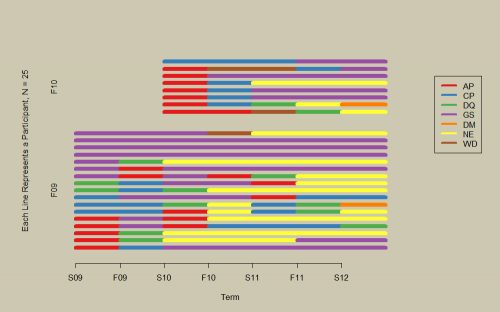
Żałuję, że nie znalazłem odpowiedzi @bdemarest przed napisaniem pakietu R, aby rozwiązać ten problem, ale ponieważ OP zażądał dodatkowych aktualizacji, podzielę się jeszcze jednym rozwiązaniem. To, co bdemarest zasugerował na rysunku 4, jest tym, co nazywam rodzajem poziomego wykresu liniowego.
Podczas opracowywania pakietu longCatEDA R odkryliśmy, że sortowanie danych było kluczowe dla tworzenia użytecznych Wykresów (zobacz example(sorter) i raport połączony w komentarzu poniżej, aby uzyskać szczegóły techniczne), zwłaszcza ze względu na rozmiar problemu stał się Duży. Na przykład rozpoczęliśmy problem z codziennymi danymi o piciu (abstynenci, zażywanie, nadużywanie) dla kilku tysięcy uczestników w ciągu 3 lat (>1000 dni).
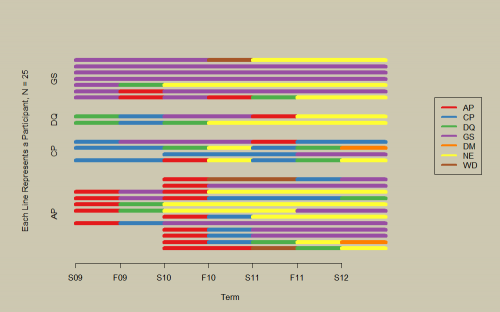
Kod do zastosowania poziomego wykresu linii do danych @ eipi10 znajduje się poniżej. Rysunek 1 stratyfikuje przez term, A Rysunek 2 stratyfikuje przez pierwszy status, jak na rysunku 4 z @bdemarest, chociaż wyniki nie są identyczne ze względu na sortowanie w obrębie warstw.
Rysunek 1
Rysunek 2
# libraries
install.packages('longCatEDA')
library(longCatEDA)
library(RColorBrewer)
# transform data long to wide
dfw <- reshape(df1,
timevar = 'term',
idvar = c('id', 'cohort'),
direction = 'wide')
# set up objects required by longCat()
y <- dfw[,seq(3,15,by=2)]
Labels <- levels(df1$standing)
tLabels <- levels(df1$term)
groupLabels <- levels(dfw$cohort)
# use the same colors as bdemarest
cols <- brewer.pal(7, "Set1")
# plot the longCat object
png('plot1.png', width=10, height=6.25, units='in', res=100)
par(bg='cornsilk3', mar=c(5.1, 4.1, 4.1, 8.1), xpd=TRUE)
lc <- longCat(y=y, Labels=Labels, tLabels=tLabels, id=dfw$id)
longCatPlot(lc, cols=cols, xlab='Term', lwd=8, legendBuffer=0)
legend(8.1, 25, legend=Labels, col=cols, lty=1, lwd=4)
dev.off()
# stratify by term
png('plot2.png', width=10, height=6.25, units='in', res=100)
par(bg='cornsilk3', mar=c(5.1, 4.1, 4.1, 8.1), xpd=TRUE)
lc.g <- sorter(lc, group=dfw$cohort, groupLabels=groupLabels)
longCatPlot(lc.g, cols=cols, xlab='Term', lwd=8, legendBuffer=0)
legend(8.1, 25, legend=Labels, col=cols, lty=1, lwd=4)
dev.off()
# stratify by first status, akin to Figure 4 by bdemarest
png('plot2.png', width=10, height=6.25, units='in', res=100)
par(bg='cornsilk3', mar=c(5.1, 4.1, 4.1, 8.1), xpd=TRUE)
first <- apply(!is.na(y), 1, function(x) which(x)[1])
first <- y[cbind(seq_along(first), first)]
lc.1 <- sorter(lc, group=factor(first), groupLabels = sort(unique(first)))
longCatPlot(lc.1, cols=cols, xlab='Term', lwd=8, legendBuffer=0)
legend(8.1, 25, legend=Labels, col=cols, lty=1, lwd=4)
dev.off()
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2017-01-24 00:37:30