Działki Side-by-side z ggplot2
Chciałbym umieścić dwie działki obok siebie za pomocą pakietu ggplot2 , czyli zrobić odpowiednik par(mfrow=c(1,2)).
Na przykład, chciałbym, aby następujące dwa wykresy pokazywały się obok siebie w tej samej skali.
x <- rnorm(100)
eps <- rnorm(100,0,.2)
qplot(x,3*x+eps)
qplot(x,2*x+eps)
qplot(displ, hwy, data=mpg, facets = . ~ year) + geom_smooth()
12 answers
Dowolne ggploty obok siebie (lub N działek na siatce)
Funkcja grid.arrange() w gridExtra pakiet będzie łączyć wiele działek; tak można umieścić dwa obok siebie.
require(gridExtra)
plot1 <- qplot(1)
plot2 <- qplot(1)
grid.arrange(plot1, plot2, ncol=2)
Jest to przydatne, gdy dwa wykresy nie są oparte na tych samych danych, na przykład, jeśli chcesz wykreślić różne zmienne bez użycia metody reshape ().
To wykreśli wynik jako efekt uboczny. Aby wydrukować efekt uboczny do pliku, określ sterownik urządzenia (np. pdf, png, itd), np.
pdf("foo.pdf")
grid.arrange(plot1, plot2)
dev.off()
Lub stosować arrangeGrob() w połączeniu z ggsave(),
ggsave("foo.pdf", arrangeGrob(plot1, plot2))
Jest to odpowiednik tworzenia dwóch odrębnych Wykresów za pomocą par(mfrow = c(1,2)). To nie tylko oszczędza czas organizowania danych, jest konieczne, gdy chcesz dwa różne wykresy.
Dodatek: Korzystanie Z Aspektów
Aspekty są pomocne w tworzeniu podobnych działek dla różnych grup. Jest to zaznaczone poniżej w wielu odpowiedziach poniżej, ale chcę podkreślić to podejście z przykładami równoważnymi do powyższych działek.
mydata <- data.frame(myGroup = c('a', 'b'), myX = c(1,1))
qplot(data = mydata,
x = myX,
facets = ~myGroup)
ggplot(data = mydata) +
geom_bar(aes(myX)) +
facet_wrap(~myGroup)
Update
Funkcja plot_grid w cowplot warto sprawdzić jako alternatywę dla grid.arrange. Zobacz answer by @claus-wilke poniżej i this vignette dla równoważnego podejścia; ale funkcja pozwala na dokładniejsze sterowanie lokalizacją i rozmiarem wykresu, na podstawie this vignette .
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2017-05-23 11:47:36
Jedną z wad rozwiązań opartych na grid.arrange jest to, że utrudniają one oznaczanie działek literami (A, B, itp.), czego wymaga większość czasopism.
Napisałem pakiet cowplot, aby rozwiązać ten (i kilka innych) problemów, w szczególności funkcję plot_grid():
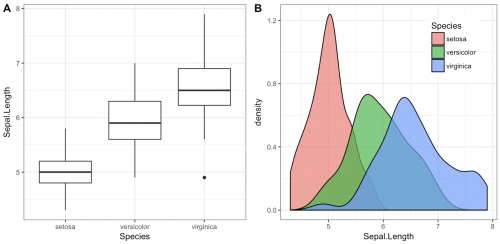
library(cowplot)
iris1 <- ggplot(iris, aes(x = Species, y = Sepal.Length)) +
geom_boxplot() + theme_bw()
iris2 <- ggplot(iris, aes(x = Sepal.Length, fill = Species)) +
geom_density(alpha = 0.7) + theme_bw() +
theme(legend.position = c(0.8, 0.8))
plot_grid(iris1, iris2, labels = "AUTO")
Obiekt, który zwraca plot_grid() jest kolejnym obiektem ggplot2 i możesz go zapisać za pomocą ggsave() jak zwykle:
p <- plot_grid(iris1, iris2, labels = "AUTO")
ggsave("plot.pdf", p)
Alternatywnie, można użyć krowiego funkcja save_plot(), która jest cienką owijką wokół ggsave(), która ułatwia uzyskanie prawidłowych wymiarów dla połączonych Wykresów, np.:
p <- plot_grid(iris1, iris2, labels = "AUTO")
save_plot("plot.pdf", p, ncol = 2)
(argument ncol = 2 mówi save_plot(), że istnieją dwie działki obok siebie, a save_plot() sprawia, że zapisany obraz jest dwukrotnie szerszy.)
Aby uzyskać bardziej szczegółowy opis jak układać działki w siatce, zobacz ta winieta. istnieje również winieta wyjaśniająca, jak zrobić fabułę z wspólną legendą.
Jeden częsty punkt zamieszania jest to, że pakiet cowplot zmienia domyślny motyw ggplot2. Pakiet zachowuje się w ten sposób, ponieważ został pierwotnie napisany dla wewnętrznych zastosowań laboratoryjnych i nigdy nie używamy domyślnego motywu. Jeśli powoduje to problemy, możesz użyć jednego z następujących trzech podejść do obejścia ich:]}
1. Ustaw motyw ręcznie dla każdej fabuły. myślę, że dobrą praktyką jest zawsze określanie konkretnego tematu dla każdej fabuły, tak jak zrobiłem to z + theme_bw() w powyższym przykładzie. Jeśli określ konkretny motyw, domyślny motyw nie ma znaczenia.
2. Przywróć domyślny motyw z powrotem do domyślnego ggplot2. możesz to zrobić za pomocą jednej linii kodu:
theme_set(theme_gray())
3. Wywołanie funkcji cowplot bez dołączania pakietu. nie można również wywoływać library(cowplot) lub require(cowplot), a zamiast tego wywoływać funkcje cowplot przez prepending cowplot::. Np. powyższy przykład użycia domyślnego motywu ggplot2 zostań:
## Commented out, we don't call this
# library(cowplot)
iris1 <- ggplot(iris, aes(x = Species, y = Sepal.Length)) +
geom_boxplot()
iris2 <- ggplot(iris, aes(x = Sepal.Length, fill = Species)) +
geom_density(alpha = 0.7) +
theme(legend.position = c(0.8, 0.8))
cowplot::plot_grid(iris1, iris2, labels = "AUTO")
Update: od ggplot2 3.0.0, działki mogą być oznaczone bezpośrednio, patrz np. tutaj.
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2018-07-28 17:06:43
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2015-08-15 20:07:40
Tak, myślę, że musisz odpowiednio zorganizować swoje dane. Jednym ze sposobów byłoby to:
X <- data.frame(x=rep(x,2),
y=c(3*x+eps, 2*x+eps),
case=rep(c("first","second"), each=100))
qplot(x, y, data=X, facets = . ~ case) + geom_smooth()
Jestem pewien, że są lepsze triki w plyr lub reshape -- nadal nie jestem na bieżąco na wszystkich tych potężnych pakietach Hadley.
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2009-08-08 18:56:06
Używając pakietu reshape możesz zrobić coś takiego.
library(ggplot2)
wide <- data.frame(x = rnorm(100), eps = rnorm(100, 0, .2))
wide$first <- with(wide, 3 * x + eps)
wide$second <- with(wide, 2 * x + eps)
long <- melt(wide, id.vars = c("x", "eps"))
ggplot(long, aes(x = x, y = value)) + geom_smooth() + geom_point() + facet_grid(.~ variable)
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2009-08-10 09:52:11
Używając pakietu patchwork , możesz po prostu użyć operatora +:
# install.packages("devtools")
devtools::install_github("thomasp85/patchwork")
library(ggplot2)
p1 <- ggplot(mtcars) + geom_point(aes(mpg, disp))
p2 <- ggplot(mtcars) + geom_boxplot(aes(gear, disp, group = gear))
library(patchwork)
p1 + p2
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2018-01-23 10:24:14
Update: ta odpowiedź jest bardzo stara. {[1] } jest teraz zalecanym podejściem. Zostawiam to tutaj na wypadek, gdyby się przydało.
Stephen Turner opublikował funkcję arrange() na Getting Genetics Done blog (Zobacz post dla Instrukcji aplikacji)
vp.layout <- function(x, y) viewport(layout.pos.row=x, layout.pos.col=y)
arrange <- function(..., nrow=NULL, ncol=NULL, as.table=FALSE) {
dots <- list(...)
n <- length(dots)
if(is.null(nrow) & is.null(ncol)) { nrow = floor(n/2) ; ncol = ceiling(n/nrow)}
if(is.null(nrow)) { nrow = ceiling(n/ncol)}
if(is.null(ncol)) { ncol = ceiling(n/nrow)}
## NOTE see n2mfrow in grDevices for possible alternative
grid.newpage()
pushViewport(viewport(layout=grid.layout(nrow,ncol) ) )
ii.p <- 1
for(ii.row in seq(1, nrow)){
ii.table.row <- ii.row
if(as.table) {ii.table.row <- nrow - ii.table.row + 1}
for(ii.col in seq(1, ncol)){
ii.table <- ii.p
if(ii.p > n) break
print(dots[[ii.table]], vp=vp.layout(ii.table.row, ii.col))
ii.p <- ii.p + 1
}
}
}
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2018-07-16 04:06:51
Ggplot2 opiera się na grafice siatki, która zapewnia inny system aranżacji działek na stronie. Polecenie par(mfrow...) nie ma bezpośredniego odpowiednika, ponieważ obiekty siatki (zwane grobs) niekoniecznie są rysowane natychmiast, ale mogą być przechowywane i manipulowane jako zwykłe obiekty R przed konwersją na wyjście graficzne. Umożliwia to większą elastyczność niż model narysuj teraz bazowego modelu grafiki, ale strategia musi być trochę inna.
I wrote grid.arrange() to provide a simple interface as close as possible to par(mfrow). W swojej najprostszej formie kod wyglądałby następująco:
library(ggplot2)
x <- rnorm(100)
eps <- rnorm(100,0,.2)
p1 <- qplot(x,3*x+eps)
p2 <- qplot(x,2*x+eps)
library(gridExtra)
grid.arrange(p1, p2, ncol = 2)
Więcej opcji znajdziesz w tej winiecie .
Jedną z powszechnych skarg jest to, że działki niekoniecznie są wyrównane, np. gdy mają etykiety osi o różnej wielkości, ale jest to z założenia: grid.arrange nie próbuje specjalnych obiektów ggplot2 i traktuje je jednakowo do innych grobów (działki kratowe, dla instancja). Umieszczono w nim jedynie groble w układzie prostokątnym.
Dla specjalnego przypadku obiektów ggplot2, napisałem inną funkcję, ggarrange, z podobnym interfejsem, która próbuje wyrównać panele wykresu (w tym wykresy) i stara się przestrzegać współczynników proporcji, gdy jest zdefiniowany przez użytkownika.
library(egg)
ggarrange(p1, p2, ncol = 2)
Obie funkcje są kompatybilne z ggsave(). Aby uzyskać ogólny przegląd różnych opcji i kontekst historyczny, ta winieta oferuje dodatkowe informacje .
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2017-12-02 04:20:12
Using tidyverse
x <- rnorm(100)
eps <- rnorm(100,0,.2)
df <- data.frame(x, eps) %>%
mutate(p1 = 3*x+eps, p2 = 2*x+eps) %>%
tidyr::gather("plot", "value", 3:4) %>%
ggplot(aes(x = x , y = value))+ geom_point()+geom_smooth()+facet_wrap(~plot, ncol =2)
df
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2018-04-12 21:52:31
Powyższe rozwiązania mogą nie być skuteczne, jeśli chcesz narysować wiele wykresów ggplot za pomocą pętli (np. jak zadano tutaj: tworzenie wielu wykresów w ggplot z różnymi wartościami osi Y Za pomocą pętli ), co jest pożądanym krokiem w analizie nieznanych (lub dużych) zestawów danych (np. gdy chcesz narysować liczby wszystkich zmiennych w zestawie danych).
Poniższy kod pokazuje, jak to zrobić za pomocą wspomnianego powyżej ' multipot ()', którego źródło znajduje się tutaj: http://www.cookbook-r.com/Graphs/Multiple_graphs_on_one_page_ (ggplot2):
plotAllCounts <- function (dt){
plots <- list();
for(i in 1:ncol(dt)) {
strX = names(dt)[i]
print(sprintf("%i: strX = %s", i, strX))
plots[[i]] <- ggplot(dt) + xlab(strX) +
geom_point(aes_string(strX),stat="count")
}
columnsToPlot <- floor(sqrt(ncol(dt)))
multiplot(plotlist = plots, cols = columnsToPlot)
}
Teraz uruchom funkcję-aby uzyskać zliczanie wszystkich zmiennych wydrukowanych za pomocą ggplot na jednej stronie
dt = ggplot2::diamonds
plotAllCounts(dt)
Należy zauważyć, że:
użycie aes(get(strX)), którego normalnie używałbyś w pętlach podczas pracy z ggplot, w powyższym kodzie zamiast aes_string(strX) nie narysuje żądanych Wykresów. Zamiast tego wykreśli ostatnią fabułę wiele razy. Nie wiem dlaczego - może trzeba zrobić aes i aes_string są wywoływane w ggplot.
W przeciwnym razie, mam nadzieję, że funkcja okaże się przydatna.
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2017-05-23 11:33:25
Jest również Pakiet multipanelfigure , który warto wspomnieć. Zobacz również tę odpowiedź .
library(ggplot2)
theme_set(theme_bw())
q1 <- ggplot(mtcars) + geom_point(aes(mpg, disp))
q2 <- ggplot(mtcars) + geom_boxplot(aes(gear, disp, group = gear))
q3 <- ggplot(mtcars) + geom_smooth(aes(disp, qsec))
q4 <- ggplot(mtcars) + geom_bar(aes(carb))
library(magrittr)
library(multipanelfigure)
figure1 <- multi_panel_figure(columns = 2, rows = 2, panel_label_type = "none")
# show the layout
figure1

figure1 %<>%
fill_panel(q1, column = 1, row = 1) %<>%
fill_panel(q2, column = 2, row = 1) %<>%
fill_panel(q3, column = 1, row = 2) %<>%
fill_panel(q4, column = 2, row = 2)
figure1

# complex layout
figure2 <- multi_panel_figure(columns = 3, rows = 3, panel_label_type = "upper-roman")
figure2

figure2 %<>%
fill_panel(q1, column = 1:2, row = 1) %<>%
fill_panel(q2, column = 3, row = 1) %<>%
fill_panel(q3, column = 1, row = 2) %<>%
fill_panel(q4, column = 2:3, row = 2:3)
figure2

Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2018-07-07 06:29:18
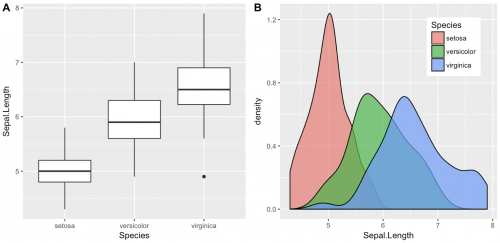
Pakiet cowplot daje Ci miły sposób, aby to zrobić, w sposób, który pasuje do publikacji.
x <- rnorm(100)
eps <- rnorm(100,0,.2)
A = qplot(x,3*x+eps, geom = c("point", "smooth"))+theme_gray()
B = qplot(x,2*x+eps, geom = c("point", "smooth"))+theme_gray()
cowplot::plot_grid(A, B, labels = c("A", "B"), align = "v")
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2017-10-20 13:42:45