Jak połączyć (scalić) ramki danych (wewnętrzne, zewnętrzne, lewe, prawe)?
Podane dwie ramki danych:
df1 = data.frame(CustomerId = c(1:6), Product = c(rep("Toaster", 3), rep("Radio", 3)))
df2 = data.frame(CustomerId = c(2, 4, 6), State = c(rep("Alabama", 2), rep("Ohio", 1)))
df1
# CustomerId Product
# 1 Toaster
# 2 Toaster
# 3 Toaster
# 4 Radio
# 5 Radio
# 6 Radio
df2
# CustomerId State
# 2 Alabama
# 4 Alabama
# 6 Ohio
Jak zrobić styl bazy danych, czyli styl sql, dołącza? Czyli jak mam:
- An INNER join of
df1anddf2:
Zwraca tylko wiersze, w których lewa tabela ma pasujące klucze w prawej tabeli. - An zewnętrzne połączenie z
df1idf2:
Zwraca wszystkie wiersze z obu tabel, Dołącz rekordy z lewej strony, które mają pasujące klucze w prawej tabeli. - A lewa zewnętrzna join (lub po prostu left join) of
df1anddf2
Zwraca wszystkie wiersze z lewej tabeli i wszystkie wiersze z pasującymi klawiszami z prawej tabeli. - A prawy zewnętrzny łącznik z
df1idf2
Zwraca wszystkie wiersze z prawej tabeli i wszystkie wiersze z pasującymi klawiszami z lewej tabeli.
Dodatkowy kredyt:
Jak mogę wykonać polecenie SQL style select?
13 answers
Za pomocą funkcji merge i jej opcjonalnych parametrów:
Złącze wewnętrzne: merge(df1, df2) będzie działać dla tych przykładów, ponieważ R automatycznie łączy ramki za pomocą wspólnych nazw zmiennych, ale najprawdopodobniej chcesz podać merge(df1, df2, by = "CustomerId"), aby upewnić się, że pasujesz tylko do żądanych pól. Możesz również użyć parametrów by.x i by.y, Jeśli pasujące zmienne mają różne nazwy w różnych ramkach danych.
zewnętrzne Dołącz: merge(x = df1, y = df2, by = "CustomerId", all = TRUE)
Lewa zewnętrzna: merge(x = df1, y = df2, by = "CustomerId", all.x = TRUE)
Prawy zewnętrzny: merge(x = df1, y = df2, by = "CustomerId", all.y = TRUE)
Cross join: merge(x = df1, y = df2, by = NULL)
podobnie jak w przypadku połączenia wewnętrznego, prawdopodobnie chciałbyś jawnie przekazać "CustomerId" do R jako zmiennej pasującej. myślę, że prawie zawsze najlepiej jest jawnie podać identyfikatory, na których chcesz się połączyć; bezpieczniej jest, jeśli Dane wejściowe.ramki zmieniają się nieoczekiwanie i łatwiej Czytaj dalej.
Można scalić na wielu kolumnach, dając by wektor, np. by = c("CustomerId", "OrderId").
Jeśli nazwy kolumn do scalenia nie są takie same, możesz podać np. by.x = "CustomerId_in_df1",by.y = "CustomerId_in_df2" where CustomerId_in_df1 is the name of the column in the first data frame and CustomerId_in_df2 ' jest nazwą kolumny w drugiej ramce danych. (Mogą to być również wektory, jeśli trzeba scalić na wielu kolumnach.)
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2018-05-04 15:43:49
Zalecałbym sprawdzenie pakietu Sqldf Gabora Grothendiecka , który pozwala wyrazić te operacje w SQL.
library(sqldf)
## inner join
df3 <- sqldf("SELECT CustomerId, Product, State
FROM df1
JOIN df2 USING(CustomerID)")
## left join (substitute 'right' for right join)
df4 <- sqldf("SELECT CustomerId, Product, State
FROM df1
LEFT JOIN df2 USING(CustomerID)")
Uważam składnię SQL za prostszą i bardziej naturalną niż jej odpowiednik R (ale może to odzwierciedlać mój błąd RDBMS).
Aby uzyskać więcej informacji na temat połączeń, Zobacz GitHub Sqldf Gabora.Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2015-06-12 07:25:09
Są dane .table podejście do połączenia wewnętrznego, które jest bardzo efektywne w czasie i pamięci (i niezbędne dla niektórych większych danych.ramki):
library(data.table)
dt1 <- data.table(df1, key = "CustomerId")
dt2 <- data.table(df2, key = "CustomerId")
joined.dt1.dt.2 <- dt1[dt2]
merge działa również na danych.tabele (jak to jest ogólne i wywołania merge.data.table)
merge(dt1, dt2)
Data.tabela udokumentowana na stackoverflow:
Jak zrobić dane.operacja scalania tabeli
tłumaczenie złączeń SQL na klucze obce na dane R.składnia tabeli
wydajne alternatywy do łączenia dla większych data.ramki R
Jak zrobić podstawowe lewe zewnętrzne połączenie z danymi.stolik w R?
Kolejną opcją jest funkcja join znaleziona w plyr pakiet
library(plyr)
join(df1, df2,
type = "inner")
# CustomerId Product State
# 1 2 Toaster Alabama
# 2 4 Radio Alabama
# 3 6 Radio Ohio
Opcje dla type: inner, left, right, full.
From ?join:merge, [join] zachowuje kolejność x bez względu na rodzaj połączenia.
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2017-06-11 07:48:14
Możesz również dołączyć za pomocą niesamowitego pakietu Hadley Wickham dplyr.
library(dplyr)
#make sure that CustomerId cols are both type numeric
#they ARE not using the provided code in question and dplyr will complain
df1$CustomerId <- as.numeric(df1$CustomerId)
df2$CustomerId <- as.numeric(df2$CustomerId)
Mutowanie łączy: dodawanie kolumn do df1 przy użyciu dopasowań w df2
#inner
inner_join(df1, df2)
#left outer
left_join(df1, df2)
#right outer
right_join(df1, df2)
#alternate right outer
left_join(df2, df1)
#full join
full_join(df1, df2)
Filtrowanie łączy: filtrowanie wierszy w df1, nie modyfikowanie kolumn
semi_join(df1, df2) #keep only observations in df1 that match in df2.
anti_join(df1, df2) #drops all observations in df1 that match in df2.
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2016-09-29 15:35:59
Jest kilka dobrych przykładów zrobienia tego na R Wiki . Ukradnę tu parę:
Metoda Merge
Ponieważ twoje klucze mają taką samą nazwę, krótką drogą do połączenia wewnętrznego jest merge ():
merge(df1,df2)
Pełne połączenie wewnętrzne (wszystkie rekordy z obu tabel) można utworzyć za pomocą słowa kluczowego "all":
merge(df1,df2, all=TRUE)
Lewy zewnętrzny łącznik df1 i df2:
merge(df1,df2, all.x=TRUE)
Prawe połączenie zewnętrzne df1 i df2:
merge(df1,df2, all.y=TRUE)
Możesz je przewrócić, uderzyć i pocierać je w dół, aby uzyskać pozostałe dwa zewnętrzne przyłącza, o które pytałeś:)
Metoda Indeksu
Lewy zewnętrzny łącznik z df1 po lewej stronie przy użyciu metody indeksu dolnego to:
df1[,"State"]<-df2[df1[ ,"Product"], "State"]
Drugą kombinację złączeń zewnętrznych można utworzyć, używając przykładu dolnego złączeń zewnętrznych (left outer join). (tak, Wiem, że to odpowiednik powiedzenia " zostawię to jako ćwiczenie dla czytelnika...")
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2009-08-19 17:51:01
Nowe w 2014 roku:
Szczególnie jeśli interesuje Cię manipulacja danymi w ogóle (w tym sortowanie, filtrowanie, podzbiór, podsumowywanie itp.), zdecydowanie powinieneś rzucić okiem na dplyr, który zawiera wiele funkcji zaprojektowanych w celu ułatwienia pracy z ramkami danych i niektórymi innymi typami baz danych. Oferuje nawet dość rozbudowany interfejs SQL, a nawet funkcję konwersji (większość) kodu SQL bezpośrednio do R.
The four joining-related funkcje w pakiecie dplyr to (cytuję):
-
inner_join(x, y, by = NULL, copy = FALSE, ...): zwraca wszystkie wiersze z x gdzie są pasujące wartości w y i wszystkie kolumny z X i y -
left_join(x, y, by = NULL, copy = FALSE, ...): zwraca wszystkie wiersze z x oraz wszystkie kolumny z X i y -
semi_join(x, y, by = NULL, copy = FALSE, ...): zwraca wszystkie wiersze z x, gdzie są pasujące wartości w y, zachowując tylko kolumny od x. -
anti_join(x, y, by = NULL, copy = FALSE, ...): zwraca wszystkie wiersze z x gdzie nie ma pasujących wartości w y, zachowując tylko kolumny z x
It ' s all tutaj bardzo szczegółowo.
Wybór kolumn można wykonać za pomocą select(df,"column"). Jeśli to nie jest wystarczająco SQL-ish dla ciebie, to jest funkcja sql(), do której możesz wprowadzić kod SQL as-is, i wykona operację, którą podałeś tak, jak pisałeś w R cały czas (więcej informacji można znaleźć w winiecie dplyr/databases ). Na przykład, jeśli zostanie poprawnie zastosowana, sql("SELECT * FROM hflights") wybierze wszystkie kolumny z tabeli dplyr " hflights "(a"tbl").
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2015-08-25 20:22:07
Aktualizacja danych.metody tabel do łączenia zestawów danych. Zobacz poniższe przykłady dla każdego typu połączenia. Istnieją dwie metody, jedna z [.data.table podczas przekazywania drugich danych.table jako pierwszy argument do podzbioru, innym sposobem jest użycie funkcji merge, która jest wysyłana do fast data.metoda tabelaryczna.
Aktualizacja na 2016-04-01-i to nie jest żart prima aprilis!
W wersji 1.9.7 danych.połączenia tabel są teraz w stanie korzystać z istniejącego indeksu, który znacznie skraca czas łączenia. poniżej kod i benchmark nie wykorzystują danych.indeksy tabel na join . Jeśli szukasz połączenia prawie w czasie rzeczywistym, powinieneś użyć danych.indeksy tabel.
df1 = data.frame(CustomerId = c(1:6), Product = c(rep("Toaster", 3), rep("Radio", 3)))
df2 = data.frame(CustomerId = c(2L, 4L, 7L), State = c(rep("Alabama", 2), rep("Ohio", 1))) # one value changed to show full outer join
library(data.table)
dt1 = as.data.table(df1)
dt2 = as.data.table(df2)
setkey(dt1, CustomerId)
setkey(dt2, CustomerId)
# right outer join keyed data.tables
dt1[dt2]
setkey(dt1, NULL)
setkey(dt2, NULL)
# right outer join unkeyed data.tables - use `on` argument
dt1[dt2, on = "CustomerId"]
# left outer join - swap dt1 with dt2
dt2[dt1, on = "CustomerId"]
# inner join - use `nomatch` argument
dt1[dt2, nomatch=0L, on = "CustomerId"]
# anti join - use `!` operator
dt1[!dt2, on = "CustomerId"]
# inner join
merge(dt1, dt2, by = "CustomerId")
# full outer join
merge(dt1, dt2, by = "CustomerId", all = TRUE)
# see ?merge.data.table arguments for other cases
Poniżej benchmark testuje base R, sqldf, dplyr i data.stolik.
Benchmark testuje nieobsługiwane / nieindeksowane zbiory danych. Możesz uzyskać jeszcze lepszą wydajność, jeśli używasz kluczy na swoich danych.tabele lub indeksy z sqldf. Base R i dplyr nie mają indeksów ani kluczy, więc nie uwzględniłem tego scenariusza w benchmarku.
Benchmark to wykonywane na zestawach danych 5M-1 wierszy, w kolumnie join znajdują się wspólne wartości 5m-2, więc każdy scenariusz (lewy, prawy, pełny, wewnętrzny) może być przetestowany i join nadal nie jest trywialny.
library(microbenchmark)
library(sqldf)
library(dplyr)
library(data.table)
n = 5e6
set.seed(123)
df1 = data.frame(x=sample(n,n-1L), y1=rnorm(n-1L))
df2 = data.frame(x=sample(n,n-1L), y2=rnorm(n-1L))
dt1 = as.data.table(df1)
dt2 = as.data.table(df2)
# inner join
microbenchmark(times = 10L,
base = merge(df1, df2, by = "x"),
sqldf = sqldf("SELECT * FROM df1 INNER JOIN df2 ON df1.x = df2.x"),
dplyr = inner_join(df1, df2, by = "x"),
data.table = dt1[dt2, nomatch = 0L, on = "x"])
#Unit: milliseconds
# expr min lq mean median uq max neval
# base 15546.0097 16083.4915 16687.117 16539.0148 17388.290 18513.216 10
# sqldf 44392.6685 44709.7128 45096.401 45067.7461 45504.376 45563.472 10
# dplyr 4124.0068 4248.7758 4281.122 4272.3619 4342.829 4411.388 10
# data.table 937.2461 946.0227 1053.411 973.0805 1214.300 1281.958 10
# left outer join
microbenchmark(times = 10L,
base = merge(df1, df2, by = "x", all.x = TRUE),
sqldf = sqldf("SELECT * FROM df1 LEFT OUTER JOIN df2 ON df1.x = df2.x"),
dplyr = left_join(df1, df2, by = c("x"="x")),
data.table = dt2[dt1, on = "x"])
#Unit: milliseconds
# expr min lq mean median uq max neval
# base 16140.791 17107.7366 17441.9538 17414.6263 17821.9035 19453.034 10
# sqldf 43656.633 44141.9186 44777.1872 44498.7191 45288.7406 47108.900 10
# dplyr 4062.153 4352.8021 4780.3221 4409.1186 4450.9301 8385.050 10
# data.table 823.218 823.5557 901.0383 837.9206 883.3292 1277.239 10
# right outer join
microbenchmark(times = 10L,
base = merge(df1, df2, by = "x", all.y = TRUE),
sqldf = sqldf("SELECT * FROM df2 LEFT OUTER JOIN df1 ON df2.x = df1.x"),
dplyr = right_join(df1, df2, by = "x"),
data.table = dt1[dt2, on = "x"])
#Unit: milliseconds
# expr min lq mean median uq max neval
# base 15821.3351 15954.9927 16347.3093 16044.3500 16621.887 17604.794 10
# sqldf 43635.5308 43761.3532 43984.3682 43969.0081 44044.461 44499.891 10
# dplyr 3936.0329 4028.1239 4102.4167 4045.0854 4219.958 4307.350 10
# data.table 820.8535 835.9101 918.5243 887.0207 1005.721 1068.919 10
# full outer join
microbenchmark(times = 10L,
base = merge(df1, df2, by = "x", all = TRUE),
#sqldf = sqldf("SELECT * FROM df1 FULL OUTER JOIN df2 ON df1.x = df2.x"), # not supported
dplyr = full_join(df1, df2, by = "x"),
data.table = merge(dt1, dt2, by = "x", all = TRUE))
#Unit: seconds
# expr min lq mean median uq max neval
# base 16.176423 16.908908 17.485457 17.364857 18.271790 18.626762 10
# dplyr 7.610498 7.666426 7.745850 7.710638 7.832125 7.951426 10
# data.table 2.052590 2.130317 2.352626 2.208913 2.470721 2.951948 10
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2016-04-01 12:57:55
Dplyr od 0.4 zaimplementował wszystkie te połączenia, w tym outer_join, ale warto zauważyć, że przez kilka pierwszych wydań nie oferował outer_join, w wyniku czego przez dłuższy czas było dużo naprawdę kiepskiego, hakerskiego kodu obejścia (można to jeszcze znaleźć w odpowiedziach so I Kaggle z tamtego okresu).
Join-related release highlights :
- Obsługa dla Typ POSIXct, strefy czasowe, duplikaty, różne poziomy czynników. Lepsze błędy i ostrzeżenia.
- nowy argument przyrostka do kontrolowania, jakie przyrostki otrzymują zduplikowane nazwy zmiennych (#1296)
- zaimplementuj złącze prawe i złącze zewnętrzne (#96)
- mutating joins, które dodają nowe zmienne do jednej tabeli z pasujących wierszy w innej. Łączniki filtrujące, które filtrują obserwacje z jednej tabeli na podstawie czy pasują do obserwacji w drugiej tabeli.
- może teraz left_join według różnych zmiennych w każdej tabeli: DF1 % > % left_join (df2, c ("var1" = "var2"))
- *_join () nie zmienia już kolejności nazw kolumn (#324)
v0.1.3 (4/2014)
- has inner_join, left_join, semi_join, anti_join
- outer_join jeszcze nie zaimplementowany, fallback to use base:: merge () (lub plyr::join ())
- didn ' t yet implemented right_join and outer_join
- Hadley wymienia tutaj inne zalety
- jedna drobna funkcja merge ma obecnie, że dplyr nie jest możliwość oddzielenia przez.x, by.kolumny y jak np. Python pandy.
Obejścia za komentarze Hadleya w tym wydanie:
- right_join (x,y) jest tym samym co left_join(y,x) pod względem wierszy, tylko kolumny będą różnymi rzędami. Łatwo działa z select (new_column_order)
- outer_join jest w zasadzie union (left_join (x, y), right_join (x, y)) - tzn. zachowuje wszystkie wiersze w obu ramkach danych.
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2018-05-31 12:18:48
Łącząc dwie ramki danych z ~1 milionem wierszy każda, jedna z 2 kolumnami, a druga z ~20, odkryłem zaskakująco, że merge(..., all.x = TRUE, all.y = TRUE) jest szybszy niż dplyr::full_join(). To jest z dplyr v0. 4
Scalanie trwa ~17 sekund, full_join trwa ~65 sekund.
Trochę jedzenia, ponieważ zazwyczaj domyślam się dplyr do zadań manipulacji.
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2015-03-31 17:26:48
W przypadku połączenia lewego z 0..*:0..1 cardinality lub połączenia prawego z 0..1:0..* cardinality możliwe jest przypisanie jednostronnych kolumn z joinee (tabela 0..1) bezpośrednio do joinee (Tabela 0..*), a tym samym uniknięcie tworzenia zupełnie nowej tabeli danych. Wymaga to dopasowania kolumn kluczowych z joinee do joinera i indeksowania+porządkowania wierszy joinera odpowiednio do przypisania.
Jeśli klucz jest pojedynczą kolumną, to można użyć jednego połączenia do match() żeby dopasować. To jest sprawa, którą omówię w tej odpowiedzi.
Oto przykład oparty na OP, z tym, że dodałem dodatkowy wiersz do {[12] } o id 7, aby sprawdzić przypadek niepasującego klucza w stolarce. Jest to efektywnie df1 LEFT join df2:
df1 <- data.frame(CustomerId=1:6,Product=c(rep('Toaster',3L),rep('Radio',3L)));
df2 <- data.frame(CustomerId=c(2L,4L,6L,7L),State=c(rep('Alabama',2L),'Ohio','Texas'));
df1[names(df2)[-1L]] <- df2[match(df1[,1L],df2[,1L]),-1L];
df1;
## CustomerId Product State
## 1 1 Toaster <NA>
## 2 2 Toaster Alabama
## 3 3 Toaster <NA>
## 4 4 Radio Alabama
## 5 5 Radio <NA>
## 6 6 Radio Ohio
W powyższym zakodowałem założenie, że kolumna kluczowa jest pierwszą kolumną obu tabel wejściowych. Argumentowałbym, że ogólnie rzecz biorąc, nie jest to nierozsądne założenie, ponieważ, jeśli masz dane.ramka z kolumną klucza, byłoby dziwne, gdyby nie została ustawiona jako pierwsza kolumna danych.kadr od początku. I zawsze możesz zmienić kolejność kolumn, aby tak było. Korzystną konsekwencją tego założenia jest to, że nazwa kolumny klucza nie musi być mocno zakodowana, chociaż przypuszczam, że to tylko zastąpienie jednego założenia innym. Zwięzłość jest kolejną zaletą indeksowania liczb całkowitych, a także prędkości. W benchmarkach poniżej będę Zmień implementację tak, aby używała indeksowania nazw łańcuchów w celu dopasowania do konkurencyjnych implementacji.
Myślę, że jest to szczególnie odpowiednie rozwiązanie, jeśli masz kilka tabel, które chcesz pozostawić dołączyć przeciwko jednej dużej tabeli. Wielokrotne przebudowywanie całej tabeli dla każdego połączenia byłoby niepotrzebne i nieefektywne.
Z drugiej strony, jeśli chcesz, aby joinee pozostał niezmieniony przez tę operację z jakiegokolwiek powodu, to tego rozwiązania nie można użyć, ponieważ modyfikuje bezpośrednio joinee. Chociaż w takim przypadku można po prostu zrobić kopię i wykonać na miejscu przypisanie(s) na kopii.
Na marginesie krótko przyjrzałem się możliwym rozwiązaniom dopasowania kluczy wielokolumnowych. Niestety, jedyne pasujące rozwiązania, które znalazłem to:
- Nieefektywne konkatenacje. np.
- nieefektywne spójniki kartezjańskie, np.
outer(df1$a,df2$a,`==`) & outer(df1$b,df2$b,`==`). - baza R
merge()i równoważna funkcje scalania oparte na pakietach, które zawsze przydzielają nową tabelę, aby zwrócić scalony wynik, a zatem nie są odpowiednie dla rozwiązania opartego na przypisywaniu w miejscu.
match(interaction(df1$a,df1$b),interaction(df2$a,df2$b)), lub ten sam pomysł z paste().
Na przykład zobacz dopasowywanie wielu kolumn w różnych ramkach danych i uzyskiwanie innych kolumn jako wyniku, dopasuj dwie kolumny do dwóch innych kolumn, dopasowanie na wielu kolumnach , a dupe tego pytania, gdzie pierwotnie wymyśliłem rozwiązanie in-place, połączyć dwa ramki danych z różną liczbą wierszy w R .
Benchmarking
Postanowiłem zrobić własny benchmarking, aby zobaczyć, jak podejście przypisania na miejscu porównuje się do innych rozwiązań, które zostały oferowane w tym pytaniu.
Kod testowy:
library(microbenchmark);
library(data.table);
library(sqldf);
library(plyr);
library(dplyr);
solSpecs <- list(
merge=list(testFuncs=list(
inner=function(df1,df2,key) merge(df1,df2,key),
left =function(df1,df2,key) merge(df1,df2,key,all.x=T),
right=function(df1,df2,key) merge(df1,df2,key,all.y=T),
full =function(df1,df2,key) merge(df1,df2,key,all=T)
)),
data.table.unkeyed=list(argSpec='data.table.unkeyed',testFuncs=list(
inner=function(dt1,dt2,key) dt1[dt2,on=key,nomatch=0L,allow.cartesian=T],
left =function(dt1,dt2,key) dt2[dt1,on=key,allow.cartesian=T],
right=function(dt1,dt2,key) dt1[dt2,on=key,allow.cartesian=T],
full =function(dt1,dt2,key) merge(dt1,dt2,key,all=T,allow.cartesian=T) ## calls merge.data.table()
)),
data.table.keyed=list(argSpec='data.table.keyed',testFuncs=list(
inner=function(dt1,dt2) dt1[dt2,nomatch=0L,allow.cartesian=T],
left =function(dt1,dt2) dt2[dt1,allow.cartesian=T],
right=function(dt1,dt2) dt1[dt2,allow.cartesian=T],
full =function(dt1,dt2) merge(dt1,dt2,all=T,allow.cartesian=T) ## calls merge.data.table()
)),
sqldf.unindexed=list(testFuncs=list( ## note: must pass connection=NULL to avoid running against the live DB connection, which would result in collisions with the residual tables from the last query upload
inner=function(df1,df2,key) sqldf(paste0('select * from df1 inner join df2 using(',paste(collapse=',',key),')'),connection=NULL),
left =function(df1,df2,key) sqldf(paste0('select * from df1 left join df2 using(',paste(collapse=',',key),')'),connection=NULL),
right=function(df1,df2,key) sqldf(paste0('select * from df2 left join df1 using(',paste(collapse=',',key),')'),connection=NULL) ## can't do right join proper, not yet supported; inverted left join is equivalent
##full =function(df1,df2,key) sqldf(paste0('select * from df1 full join df2 using(',paste(collapse=',',key),')'),connection=NULL) ## can't do full join proper, not yet supported; possible to hack it with a union of left joins, but too unreasonable to include in testing
)),
sqldf.indexed=list(testFuncs=list( ## important: requires an active DB connection with preindexed main.df1 and main.df2 ready to go; arguments are actually ignored
inner=function(df1,df2,key) sqldf(paste0('select * from main.df1 inner join main.df2 using(',paste(collapse=',',key),')')),
left =function(df1,df2,key) sqldf(paste0('select * from main.df1 left join main.df2 using(',paste(collapse=',',key),')')),
right=function(df1,df2,key) sqldf(paste0('select * from main.df2 left join main.df1 using(',paste(collapse=',',key),')')) ## can't do right join proper, not yet supported; inverted left join is equivalent
##full =function(df1,df2,key) sqldf(paste0('select * from main.df1 full join main.df2 using(',paste(collapse=',',key),')')) ## can't do full join proper, not yet supported; possible to hack it with a union of left joins, but too unreasonable to include in testing
)),
plyr=list(testFuncs=list(
inner=function(df1,df2,key) join(df1,df2,key,'inner'),
left =function(df1,df2,key) join(df1,df2,key,'left'),
right=function(df1,df2,key) join(df1,df2,key,'right'),
full =function(df1,df2,key) join(df1,df2,key,'full')
)),
dplyr=list(testFuncs=list(
inner=function(df1,df2,key) inner_join(df1,df2,key),
left =function(df1,df2,key) left_join(df1,df2,key),
right=function(df1,df2,key) right_join(df1,df2,key),
full =function(df1,df2,key) full_join(df1,df2,key)
)),
in.place=list(testFuncs=list(
left =function(df1,df2,key) { cns <- setdiff(names(df2),key); df1[cns] <- df2[match(df1[,key],df2[,key]),cns]; df1; },
right=function(df1,df2,key) { cns <- setdiff(names(df1),key); df2[cns] <- df1[match(df2[,key],df1[,key]),cns]; df2; }
))
);
getSolTypes <- function() names(solSpecs);
getJoinTypes <- function() unique(unlist(lapply(solSpecs,function(x) names(x$testFuncs))));
getArgSpec <- function(argSpecs,key=NULL) if (is.null(key)) argSpecs$default else argSpecs[[key]];
initSqldf <- function() {
sqldf(); ## creates sqlite connection on first run, cleans up and closes existing connection otherwise
if (exists('sqldfInitFlag',envir=globalenv(),inherits=F) && sqldfInitFlag) { ## false only on first run
sqldf(); ## creates a new connection
} else {
assign('sqldfInitFlag',T,envir=globalenv()); ## set to true for the one and only time
}; ## end if
invisible();
}; ## end initSqldf()
setUpBenchmarkCall <- function(argSpecs,joinType,solTypes=getSolTypes(),env=parent.frame()) {
## builds and returns a list of expressions suitable for passing to the list argument of microbenchmark(), and assigns variables to resolve symbol references in those expressions
callExpressions <- list();
nms <- character();
for (solType in solTypes) {
testFunc <- solSpecs[[solType]]$testFuncs[[joinType]];
if (is.null(testFunc)) next; ## this join type is not defined for this solution type
testFuncName <- paste0('tf.',solType);
assign(testFuncName,testFunc,envir=env);
argSpecKey <- solSpecs[[solType]]$argSpec;
argSpec <- getArgSpec(argSpecs,argSpecKey);
argList <- setNames(nm=names(argSpec$args),vector('list',length(argSpec$args)));
for (i in seq_along(argSpec$args)) {
argName <- paste0('tfa.',argSpecKey,i);
assign(argName,argSpec$args[[i]],envir=env);
argList[[i]] <- if (i%in%argSpec$copySpec) call('copy',as.symbol(argName)) else as.symbol(argName);
}; ## end for
callExpressions[[length(callExpressions)+1L]] <- do.call(call,c(list(testFuncName),argList),quote=T);
nms[length(nms)+1L] <- solType;
}; ## end for
names(callExpressions) <- nms;
callExpressions;
}; ## end setUpBenchmarkCall()
harmonize <- function(res) {
res <- as.data.frame(res); ## coerce to data.frame
for (ci in which(sapply(res,is.factor))) res[[ci]] <- as.character(res[[ci]]); ## coerce factor columns to character
for (ci in which(sapply(res,is.logical))) res[[ci]] <- as.integer(res[[ci]]); ## coerce logical columns to integer (works around sqldf quirk of munging logicals to integers)
##for (ci in which(sapply(res,inherits,'POSIXct'))) res[[ci]] <- as.double(res[[ci]]); ## coerce POSIXct columns to double (works around sqldf quirk of losing POSIXct class) ----- POSIXct doesn't work at all in sqldf.indexed
res <- res[order(names(res))]; ## order columns
res <- res[do.call(order,res),]; ## order rows
res;
}; ## end harmonize()
checkIdentical <- function(argSpecs,solTypes=getSolTypes()) {
for (joinType in getJoinTypes()) {
callExpressions <- setUpBenchmarkCall(argSpecs,joinType,solTypes);
if (length(callExpressions)<2L) next;
ex <- harmonize(eval(callExpressions[[1L]]));
for (i in seq(2L,len=length(callExpressions)-1L)) {
y <- harmonize(eval(callExpressions[[i]]));
if (!isTRUE(all.equal(ex,y,check.attributes=F))) {
ex <<- ex;
y <<- y;
solType <- names(callExpressions)[i];
stop(paste0('non-identical: ',solType,' ',joinType,'.'));
}; ## end if
}; ## end for
}; ## end for
invisible();
}; ## end checkIdentical()
testJoinType <- function(argSpecs,joinType,solTypes=getSolTypes(),metric=NULL,times=100L) {
callExpressions <- setUpBenchmarkCall(argSpecs,joinType,solTypes);
bm <- microbenchmark(list=callExpressions,times=times);
if (is.null(metric)) return(bm);
bm <- summary(bm);
res <- setNames(nm=names(callExpressions),bm[[metric]]);
attr(res,'unit') <- attr(bm,'unit');
res;
}; ## end testJoinType()
testAllJoinTypes <- function(argSpecs,solTypes=getSolTypes(),metric=NULL,times=100L) {
joinTypes <- getJoinTypes();
resList <- setNames(nm=joinTypes,lapply(joinTypes,function(joinType) testJoinType(argSpecs,joinType,solTypes,metric,times)));
if (is.null(metric)) return(resList);
units <- unname(unlist(lapply(resList,attr,'unit')));
res <- do.call(data.frame,c(list(join=joinTypes),setNames(nm=solTypes,rep(list(rep(NA_real_,length(joinTypes))),length(solTypes))),list(unit=units,stringsAsFactors=F)));
for (i in seq_along(resList)) res[i,match(names(resList[[i]]),names(res))] <- resList[[i]];
res;
}; ## end testAllJoinTypes()
testGrid <- function(makeArgSpecsFunc,sizes,overlaps,solTypes=getSolTypes(),joinTypes=getJoinTypes(),metric='median',times=100L) {
res <- expand.grid(size=sizes,overlap=overlaps,joinType=joinTypes,stringsAsFactors=F);
res[solTypes] <- NA_real_;
res$unit <- NA_character_;
for (ri in seq_len(nrow(res))) {
size <- res$size[ri];
overlap <- res$overlap[ri];
joinType <- res$joinType[ri];
argSpecs <- makeArgSpecsFunc(size,overlap);
checkIdentical(argSpecs,solTypes);
cur <- testJoinType(argSpecs,joinType,solTypes,metric,times);
res[ri,match(names(cur),names(res))] <- cur;
res$unit[ri] <- attr(cur,'unit');
}; ## end for
res;
}; ## end testGrid()
Oto benchmark przykładu opartego na OP, który zademonstrowałem wcześniej:]}
## OP's example, supplemented with a non-matching row in df2
argSpecs <- list(
default=list(copySpec=1:2,args=list(
df1 <- data.frame(CustomerId=1:6,Product=c(rep('Toaster',3L),rep('Radio',3L))),
df2 <- data.frame(CustomerId=c(2L,4L,6L,7L),State=c(rep('Alabama',2L),'Ohio','Texas')),
'CustomerId'
)),
data.table.unkeyed=list(copySpec=1:2,args=list(
as.data.table(df1),
as.data.table(df2),
'CustomerId'
)),
data.table.keyed=list(copySpec=1:2,args=list(
setkey(as.data.table(df1),CustomerId),
setkey(as.data.table(df2),CustomerId)
))
);
## prepare sqldf
initSqldf();
sqldf('create index df1_key on df1(CustomerId);'); ## upload and create an sqlite index on df1
sqldf('create index df2_key on df2(CustomerId);'); ## upload and create an sqlite index on df2
checkIdentical(argSpecs);
testAllJoinTypes(argSpecs,metric='median');
## join merge data.table.unkeyed data.table.keyed sqldf.unindexed sqldf.indexed plyr dplyr in.place unit
## 1 inner 644.259 861.9345 923.516 9157.752 1580.390 959.2250 270.9190 NA microseconds
## 2 left 713.539 888.0205 910.045 8820.334 1529.714 968.4195 270.9185 224.3045 microseconds
## 3 right 1221.804 909.1900 923.944 8930.668 1533.135 1063.7860 269.8495 218.1035 microseconds
## 4 full 1302.203 3107.5380 3184.729 NA NA 1593.6475 270.7055 NA microseconds
Tutaj porównuję losowe Dane wejściowe, testując różne skale i różne wzory klawiszy nakładających się na dwie tabele wejściowe. Ten benchmark jest nadal ograniczony do przypadku jednokolumnowego klucza całkowitego. Ponadto, aby zapewnić, że rozwiązanie in-place będzie działać zarówno dla lewego, jak i prawego połączenia tych samych tabel, wszystkie losowe Dane testowe wykorzystują 0..1:0..1 cardinality. Jest to realizowane przez pobieranie próbek bez wymiany kolumny kluczowej pierwszych danych.ramka podczas generowania kolumny kluczowej drugich danych.rama.
makeArgSpecs.singleIntegerKey.optionalOneToOne <- function(size,overlap) {
com <- as.integer(size*overlap);
argSpecs <- list(
default=list(copySpec=1:2,args=list(
df1 <- data.frame(id=sample(size),y1=rnorm(size),y2=rnorm(size)),
df2 <- data.frame(id=sample(c(if (com>0L) sample(df1$id,com) else integer(),seq(size+1L,len=size-com))),y3=rnorm(size),y4=rnorm(size)),
'id'
)),
data.table.unkeyed=list(copySpec=1:2,args=list(
as.data.table(df1),
as.data.table(df2),
'id'
)),
data.table.keyed=list(copySpec=1:2,args=list(
setkey(as.data.table(df1),id),
setkey(as.data.table(df2),id)
))
);
## prepare sqldf
initSqldf();
sqldf('create index df1_key on df1(id);'); ## upload and create an sqlite index on df1
sqldf('create index df2_key on df2(id);'); ## upload and create an sqlite index on df2
argSpecs;
}; ## end makeArgSpecs.singleIntegerKey.optionalOneToOne()
## cross of various input sizes and key overlaps
sizes <- c(1e1L,1e3L,1e6L);
overlaps <- c(0.99,0.5,0.01);
system.time({ res <- testGrid(makeArgSpecs.singleIntegerKey.optionalOneToOne,sizes,overlaps); });
## user system elapsed
## 22024.65 12308.63 34493.19
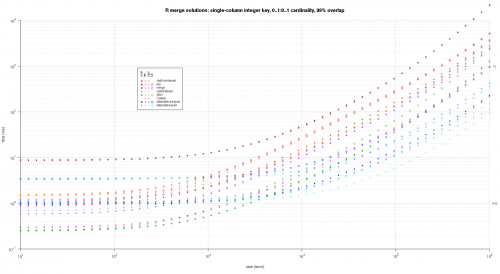
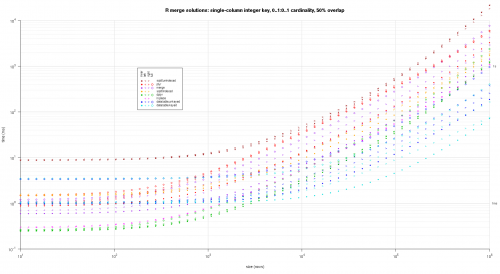
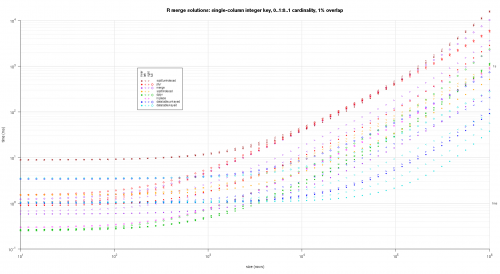
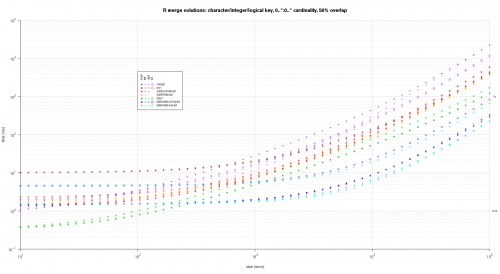
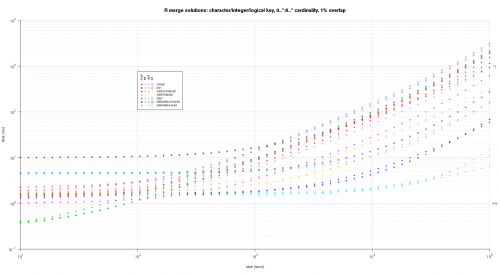
Napisałem jakiś kod do tworzenia log-log Wykresów z powyższe wyniki. Wygenerowałem osobny wykres dla każdego procentu nakładania się. Jest to trochę bałagan, ale lubię mieć wszystkie typy rozwiązań i typy połączeń reprezentowane na tej samej działce.
Użyłem interpolacji spline, aby pokazać gładką krzywą dla każdej kombinacji typu rozwiązanie / połączenie, narysowanej za pomocą poszczególnych symboli pch. Typ połączenia jest rejestrowany przez symbol pch, używając kropki dla nawiasów wewnętrznych, lewego i prawego dla lewego i prawego oraz diamentu dla pełnego. Typ rozwiązania jest przechwytywany według koloru pokazanego w legendzie.
plotRes <- function(res,titleFunc,useFloor=F) {
solTypes <- setdiff(names(res),c('size','overlap','joinType','unit')); ## derive from res
normMult <- c(microseconds=1e-3,milliseconds=1); ## normalize to milliseconds
joinTypes <- getJoinTypes();
cols <- c(merge='purple',data.table.unkeyed='blue',data.table.keyed='#00DDDD',sqldf.unindexed='brown',sqldf.indexed='orange',plyr='red',dplyr='#00BB00',in.place='magenta');
pchs <- list(inner=20L,left='<',right='>',full=23L);
cexs <- c(inner=0.7,left=1,right=1,full=0.7);
NP <- 60L;
ord <- order(decreasing=T,colMeans(res[res$size==max(res$size),solTypes],na.rm=T));
ymajors <- data.frame(y=c(1,1e3),label=c('1ms','1s'),stringsAsFactors=F);
for (overlap in unique(res$overlap)) {
x1 <- res[res$overlap==overlap,];
x1[solTypes] <- x1[solTypes]*normMult[x1$unit]; x1$unit <- NULL;
xlim <- c(1e1,max(x1$size));
xticks <- 10^seq(log10(xlim[1L]),log10(xlim[2L]));
ylim <- c(1e-1,10^((if (useFloor) floor else ceiling)(log10(max(x1[solTypes],na.rm=T))))); ## use floor() to zoom in a little more, only sqldf.unindexed will break above, but xpd=NA will keep it visible
yticks <- 10^seq(log10(ylim[1L]),log10(ylim[2L]));
yticks.minor <- rep(yticks[-length(yticks)],each=9L)*1:9;
plot(NA,xlim=xlim,ylim=ylim,xaxs='i',yaxs='i',axes=F,xlab='size (rows)',ylab='time (ms)',log='xy');
abline(v=xticks,col='lightgrey');
abline(h=yticks.minor,col='lightgrey',lty=3L);
abline(h=yticks,col='lightgrey');
axis(1L,xticks,parse(text=sprintf('10^%d',as.integer(log10(xticks)))));
axis(2L,yticks,parse(text=sprintf('10^%d',as.integer(log10(yticks)))),las=1L);
axis(4L,ymajors$y,ymajors$label,las=1L,tick=F,cex.axis=0.7,hadj=0.5);
for (joinType in rev(joinTypes)) { ## reverse to draw full first, since it's larger and would be more obtrusive if drawn last
x2 <- x1[x1$joinType==joinType,];
for (solType in solTypes) {
if (any(!is.na(x2[[solType]]))) {
xy <- spline(x2$size,x2[[solType]],xout=10^(seq(log10(x2$size[1L]),log10(x2$size[nrow(x2)]),len=NP)));
points(xy$x,xy$y,pch=pchs[[joinType]],col=cols[solType],cex=cexs[joinType],xpd=NA);
}; ## end if
}; ## end for
}; ## end for
## custom legend
## due to logarithmic skew, must do all distance calcs in inches, and convert to user coords afterward
## the bottom-left corner of the legend will be defined in normalized figure coords, although we can convert to inches immediately
leg.cex <- 0.7;
leg.x.in <- grconvertX(0.275,'nfc','in');
leg.y.in <- grconvertY(0.6,'nfc','in');
leg.x.user <- grconvertX(leg.x.in,'in');
leg.y.user <- grconvertY(leg.y.in,'in');
leg.outpad.w.in <- 0.1;
leg.outpad.h.in <- 0.1;
leg.midpad.w.in <- 0.1;
leg.midpad.h.in <- 0.1;
leg.sol.w.in <- max(strwidth(solTypes,'in',leg.cex));
leg.sol.h.in <- max(strheight(solTypes,'in',leg.cex))*1.5; ## multiplication factor for greater line height
leg.join.w.in <- max(strheight(joinTypes,'in',leg.cex))*1.5; ## ditto
leg.join.h.in <- max(strwidth(joinTypes,'in',leg.cex));
leg.main.w.in <- leg.join.w.in*length(joinTypes);
leg.main.h.in <- leg.sol.h.in*length(solTypes);
leg.x2.user <- grconvertX(leg.x.in+leg.outpad.w.in*2+leg.main.w.in+leg.midpad.w.in+leg.sol.w.in,'in');
leg.y2.user <- grconvertY(leg.y.in+leg.outpad.h.in*2+leg.main.h.in+leg.midpad.h.in+leg.join.h.in,'in');
leg.cols.x.user <- grconvertX(leg.x.in+leg.outpad.w.in+leg.join.w.in*(0.5+seq(0L,length(joinTypes)-1L)),'in');
leg.lines.y.user <- grconvertY(leg.y.in+leg.outpad.h.in+leg.main.h.in-leg.sol.h.in*(0.5+seq(0L,length(solTypes)-1L)),'in');
leg.sol.x.user <- grconvertX(leg.x.in+leg.outpad.w.in+leg.main.w.in+leg.midpad.w.in,'in');
leg.join.y.user <- grconvertY(leg.y.in+leg.outpad.h.in+leg.main.h.in+leg.midpad.h.in,'in');
rect(leg.x.user,leg.y.user,leg.x2.user,leg.y2.user,col='white');
text(leg.sol.x.user,leg.lines.y.user,solTypes[ord],cex=leg.cex,pos=4L,offset=0);
text(leg.cols.x.user,leg.join.y.user,joinTypes,cex=leg.cex,pos=4L,offset=0,srt=90); ## srt rotation applies *after* pos/offset positioning
for (i in seq_along(joinTypes)) {
joinType <- joinTypes[i];
points(rep(leg.cols.x.user[i],length(solTypes)),ifelse(colSums(!is.na(x1[x1$joinType==joinType,solTypes[ord]]))==0L,NA,leg.lines.y.user),pch=pchs[[joinType]],col=cols[solTypes[ord]]);
}; ## end for
title(titleFunc(overlap));
readline(sprintf('overlap %.02f',overlap));
}; ## end for
}; ## end plotRes()
titleFunc <- function(overlap) sprintf('R merge solutions: single-column integer key, 0..1:0..1 cardinality, %d%% overlap',as.integer(overlap*100));
plotRes(res,titleFunc,T);
Oto drugi duży benchmark, który jest bardziej wytrzymały, pod względem liczby i rodzajów kolumn kluczowych, a także kardynalności. W tym benchmarku używam trzech kolumn kluczowych: jednego znaku, jednej liczby całkowitej i jednej logicznej, bez ograniczeń kardynalności (tj. {20]}). (Generalnie nie jest wskazane definiowanie kolumn kluczowych z podwójnymi lub złożonymi wartościami z powodu komplikacji w porównaniu zmiennoprzecinkowym i zasadniczo nikt nigdy nie używa typu raw, a tym bardziej dla kolumn kluczowych, więc nie uwzględniłem tych typów w kolumnach kluczy. Ponadto, dla dobra informacji, początkowo próbowałem użyć czterech kolumn kluczowych, włączając kolumnę klucza POSIXct, ale Typ POSIXct nie grał dobrze z rozwiązaniem sqldf.indexed z jakiegoś powodu, prawdopodobnie z powodu anomalii porównawczych zmiennoprzecinkowych, więc go usunąłem.)
makeArgSpecs.assortedKey.optionalManyToMany <- function(size,overlap,uniquePct=75) {
## number of unique keys in df1
u1Size <- as.integer(size*uniquePct/100);
## (roughly) divide u1Size into bases, so we can use expand.grid() to produce the required number of unique key values with repetitions within individual key columns
## use ceiling() to ensure we cover u1Size; will truncate afterward
u1SizePerKeyColumn <- as.integer(ceiling(u1Size^(1/3)));
## generate the unique key values for df1
keys1 <- expand.grid(stringsAsFactors=F,
idCharacter=replicate(u1SizePerKeyColumn,paste(collapse='',sample(letters,sample(4:12,1L),T))),
idInteger=sample(u1SizePerKeyColumn),
idLogical=sample(c(F,T),u1SizePerKeyColumn,T)
##idPOSIXct=as.POSIXct('2016-01-01 00:00:00','UTC')+sample(u1SizePerKeyColumn)
)[seq_len(u1Size),];
## rbind some repetitions of the unique keys; this will prepare one side of the many-to-many relationship
## also scramble the order afterward
keys1 <- rbind(keys1,keys1[sample(nrow(keys1),size-u1Size,T),])[sample(size),];
## common and unilateral key counts
com <- as.integer(size*overlap);
uni <- size-com;
## generate some unilateral keys for df2 by synthesizing outside of the idInteger range of df1
keys2 <- data.frame(stringsAsFactors=F,
idCharacter=replicate(uni,paste(collapse='',sample(letters,sample(4:12,1L),T))),
idInteger=u1SizePerKeyColumn+sample(uni),
idLogical=sample(c(F,T),uni,T)
##idPOSIXct=as.POSIXct('2016-01-01 00:00:00','UTC')+u1SizePerKeyColumn+sample(uni)
);
## rbind random keys from df1; this will complete the many-to-many relationship
## also scramble the order afterward
keys2 <- rbind(keys2,keys1[sample(nrow(keys1),com,T),])[sample(size),];
##keyNames <- c('idCharacter','idInteger','idLogical','idPOSIXct');
keyNames <- c('idCharacter','idInteger','idLogical');
## note: was going to use raw and complex type for two of the non-key columns, but data.table doesn't seem to fully support them
argSpecs <- list(
default=list(copySpec=1:2,args=list(
df1 <- cbind(stringsAsFactors=F,keys1,y1=sample(c(F,T),size,T),y2=sample(size),y3=rnorm(size),y4=replicate(size,paste(collapse='',sample(letters,sample(4:12,1L),T)))),
df2 <- cbind(stringsAsFactors=F,keys2,y5=sample(c(F,T),size,T),y6=sample(size),y7=rnorm(size),y8=replicate(size,paste(collapse='',sample(letters,sample(4:12,1L),T)))),
keyNames
)),
data.table.unkeyed=list(copySpec=1:2,args=list(
as.data.table(df1),
as.data.table(df2),
keyNames
)),
data.table.keyed=list(copySpec=1:2,args=list(
setkeyv(as.data.table(df1),keyNames),
setkeyv(as.data.table(df2),keyNames)
))
);
## prepare sqldf
initSqldf();
sqldf(paste0('create index df1_key on df1(',paste(collapse=',',keyNames),');')); ## upload and create an sqlite index on df1
sqldf(paste0('create index df2_key on df2(',paste(collapse=',',keyNames),');')); ## upload and create an sqlite index on df2
argSpecs;
}; ## end makeArgSpecs.assortedKey.optionalManyToMany()
sizes <- c(1e1L,1e3L,1e5L); ## 1e5L instead of 1e6L to respect more heavy-duty inputs
overlaps <- c(0.99,0.5,0.01);
solTypes <- setdiff(getSolTypes(),'in.place');
system.time({ res <- testGrid(makeArgSpecs.assortedKey.optionalManyToMany,sizes,overlaps,solTypes); });
## user system elapsed
## 38895.50 784.19 39745.53
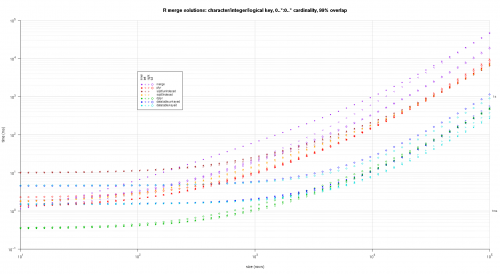
Wynik wykresy, używając tego samego kodu wykresu podanego powyżej:
titleFunc <- function(overlap) sprintf('R merge solutions: character/integer/logical key, 0..*:0..* cardinality, %d%% overlap',as.integer(overlap*100));
plotRes(res,titleFunc,F);
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2017-05-23 11:55:03
- za pomocą funkcji
mergemożemy wybrać zmienną lewej tabeli lub prawej tabeli, tak jak wszyscy znamy instrukcję select w SQL (NP: Select a.*...lub wybierz b. * z.....) -
Musimy dodać dodatkowy kod, który zostanie pobrany z nowo dołączonej tabeli .
SQL :-
select a.* from df1 a inner join df2 b on a.CustomerId=b.CustomerIdR :-
merge(df1, df2, by.x = "CustomerId", by.y = "CustomerId")[,names(df1)]
W ten sam sposób
SQL :-
select b.* from df1 a inner join df2 b on a.CustomerId=b.CustomerIdR :-
merge(df1, df2, by.x = "CustomerId", by.y = "CustomerId")[,names(df2)]
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2018-01-19 06:12:10
Dla połączenia wewnętrznego we wszystkich kolumnach, możesz również użyć fintersectz danych .table - package or intersect from the dplyr - package as an alternative to merge without specifying the by - columns. to da wiersze, które są równe między dwoma ramkami danych:
merge(df1, df2)
# V1 V2
# 1 B 2
# 2 C 3
dplyr::intersect(df1, df2)
# V1 V2
# 1 B 2
# 2 C 3
data.table::fintersect(setDT(df1), setDT(df2))
# V1 V2
# 1: B 2
# 2: C 3
Przykładowe dane:
df1 <- data.frame(V1 = LETTERS[1:4], V2 = 1:4)
df2 <- data.frame(V1 = LETTERS[2:3], V2 = 2:3)
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2017-11-02 03:48:34
/ align = "left" / innym ważnym połączeniem w stylu SQL jest " update join ", gdzie kolumny w jednej tabeli są aktualizowane (lub tworzone) przy użyciu innej tabeli.
Modyfikowanie przykładowych tabel OP...
sales = data.frame(
CustomerId = c(1, 1, 1, 3, 4, 6),
Year = 2000:2005,
Product = c(rep("Toaster", 3), rep("Radio", 3))
)
cust = data.frame(
CustomerId = c(1, 1, 4, 6),
Year = c(2001L, 2002L, 2002L, 2002L),
State = state.name[1:4]
)
sales
# CustomerId Year Product
# 1 2000 Toaster
# 1 2001 Toaster
# 1 2002 Toaster
# 3 2003 Radio
# 4 2004 Radio
# 6 2005 Radio
cust
# CustomerId Year State
# 1 2001 Alabama
# 1 2002 Alaska
# 4 2002 Arizona
# 6 2002 Arkansas
Załóżmy, że chcemy dodać stan klienta z cust do tabeli zakupów, sales, ignorując kolumnę year. Za pomocą bazy R możemy zidentyfikować pasujące wiersze, a następnie skopiować wartości nad:
sales$State <- cust$State[ match(sales$CustomerId, cust$CustomerId) ]
# CustomerId Year Product State
# 1 2000 Toaster Alabama
# 1 2001 Toaster Alabama
# 1 2002 Toaster Alabama
# 3 2003 Radio <NA>
# 4 2004 Radio Arizona
# 6 2005 Radio Arkansas
# cleanup for the next example
sales$State <- NULL
Jak widać tutaj, match wybiera pierwsze dopasowanie wiersz z tabeli klienta.
Aktualizacja połącz z wieloma kolumnami. powyższe podejście działa dobrze, gdy łączymy się tylko z jedną kolumną i jesteśmy zadowoleni z pierwszego meczu. Załóżmy, że chcemy, aby Rok pomiaru w tabeli klienta był zgodny z rokiem sprzedaży.
Jak wspomina odpowiedź @ bgoldst, match z interaction może być opcją w tym przypadku. Bardziej prosto, można użyć danych.Tabela:
library(data.table)
setDT(sales); setDT(cust)
sales[, State := cust[sales, on=.(CustomerId, Year), x.State]]
# CustomerId Year Product State
# 1: 1 2000 Toaster <NA>
# 2: 1 2001 Toaster Alabama
# 3: 1 2002 Toaster Alaska
# 4: 3 2003 Radio <NA>
# 5: 4 2004 Radio <NA>
# 6: 6 2005 Radio <NA>
# cleanup for next example
sales[, State := NULL]
Rolling update Dołącz. alternatywnie, możemy chcieć wziąć ostatni stan, w którym Klient został znaleziony:
sales[, State := cust[sales, on=.(CustomerId, Year), roll=TRUE, x.State]]
# CustomerId Year Product State
# 1: 1 2000 Toaster <NA>
# 2: 1 2001 Toaster Alabama
# 3: 1 2002 Toaster Alaska
# 4: 3 2003 Radio <NA>
# 5: 4 2004 Radio Arizona
# 6: 6 2005 Radio Arkansas
Trzy przykłady skupiają się przede wszystkim na tworzeniu/dodawaniu nowej kolumny. Zobacz powiązane R FAQ aby uzyskać przykład aktualizacji / modyfikacji istniejącej kolumny.
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2018-09-04 16:37:23