Orientacja diagonalnych etykiet na osi x w heatmapie (s)
[18]}Tworzenie heatmap w R było tematem wielu postów, dyskusji i iteracji. Moim głównym problemem jest to, że trudno jest połączyć wizualną elastyczność rozwiązań dostępnych w kratce levelplot() lub podstawowej grafice image(), z łatwym klastrem basic' s heatmap(), pheatmap 's pheatmap() lub gplots' heatmap.2(). To drobny szczegół, który chcę zmienić-orientacja diagonalna etykiet na osi X. Pokażę Ci mój punkt w kodzie.
#example data
d <- matrix(rnorm(25), 5, 5)
colnames(d) = paste("bip", 1:5, sep = "")
rownames(d) = paste("blob", 1:5, sep = "")
Można zmienić orientację na diagonalną łatwo z levelplot():
require(lattice)
levelplot(d, scale=list(x=list(rot=45)))
heatmap(d)
I tak jest tutaj:
require(gplots)
heatmap.2(d, key=F)
I na koniec mój ulubiony:
require(pheatmap)
pheatmap(d)
Ale wszystkie nie mają opcji obracania etykiet. Podręcznik dla pheatmap sugeruje, że mogę użyć grid.text do niestandardowego orientowania moich etykiet. Co za radość - szczególnie przy grupowaniu i zmianie kolejności wyświetlanych etykiet. Chyba, że coś mi umyka...
Wreszcie jest stare dobre image(). Mogę obracać etykiety, ogólnie jest to najbardziej konfigurowalne rozwiązanie, ale nie ma opcji grupowania.
image(1:nrow(d),1:ncol(d), d, axes=F, ylab="", xlab="")
text(1:ncol(d), 0, srt = 45, labels = rownames(d), xpd = TRUE)
axis(1, label=F)
axis(2, 1:nrow(d), colnames(d), las=1)
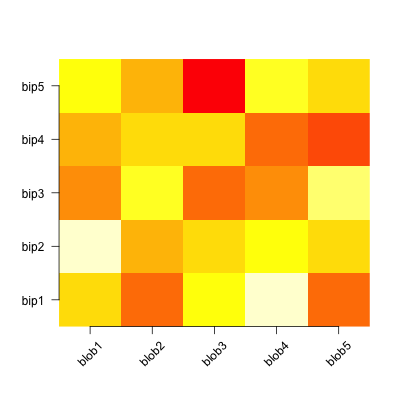
Więc co zrobić, aby uzyskać idealną, szybką heatmapę, z grupowaniem i orientacją i ładnymi funkcjami wizualnymi? Moja najlepsza oferta to zmiana heatmap() lub pheatmap(), ponieważ te dwie wydają się być najbardziej wszechstronne w dostosowaniu. Ale wszelkie rozwiązania mile widziane.
6 answers
Aby naprawić pheatmap, wszystko, co naprawdę chcesz zrobić, to przejść do pheatmap:::draw_colnames i dostosować kilka ustawień w wywołaniu do grid.text(). Jest na to jeden sposób, używając assignInNamespace(). (Może wymagać dodatkowych poprawek, ale masz zdjęcie ;):
library(grid) ## Need to attach (and not just load) grid package
library(pheatmap)
## Your data
d <- matrix(rnorm(25), 5, 5)
colnames(d) = paste("bip", 1:5, sep = "")
rownames(d) = paste("blob", 1:5, sep = "")
## Edit body of pheatmap:::draw_colnames, customizing it to your liking
draw_colnames_45 <- function (coln, ...) {
m = length(coln)
x = (1:m)/m - 1/2/m
grid.text(coln, x = x, y = unit(0.96, "npc"), vjust = .5,
hjust = 1, rot = 45, gp = gpar(...)) ## Was 'hjust=0' and 'rot=270'
}
## For pheatmap_1.0.8 and later:
draw_colnames_45 <- function (coln, gaps, ...) {
coord = pheatmap:::find_coordinates(length(coln), gaps)
x = coord$coord - 0.5 * coord$size
res = textGrob(coln, x = x, y = unit(1, "npc") - unit(3,"bigpts"), vjust = 0.5, hjust = 1, rot = 45, gp = gpar(...))
return(res)}
## 'Overwrite' default draw_colnames with your own version
assignInNamespace(x="draw_colnames", value="draw_colnames_45",
ns=asNamespace("pheatmap"))
## Try it out
pheatmap(d)
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2016-11-10 17:55:32
Jest to nieco bardziej skomplikowane niż mój komentarz zakładał, ponieważ heatmap rozbija obszar wykresu w celu narysowania dendrogramów, a ostatni obszar wykresu nie jest image wykresem, do którego chcesz dołączyć etykiety.
Istnieje rozwiązanie, ponieważ heatmap dostarcza argumentu add.expr, który pobiera wyrażenie, które ma być oceniane podczas rysowania image. Trzeba również wiedzieć, Zmiana kolejności etykiet, która odbywa się ze względu na kolejność dendrogramu. Ostatni kawałek wiąże się z odrobiną nieeleganckiego włamania, ponieważ najpierw narysuję heatmapę, aby uzyskać informacje o zmianie kolejności, a następnie użyj tego, aby narysować heatmap poprawnie z etykietami pod kątem.
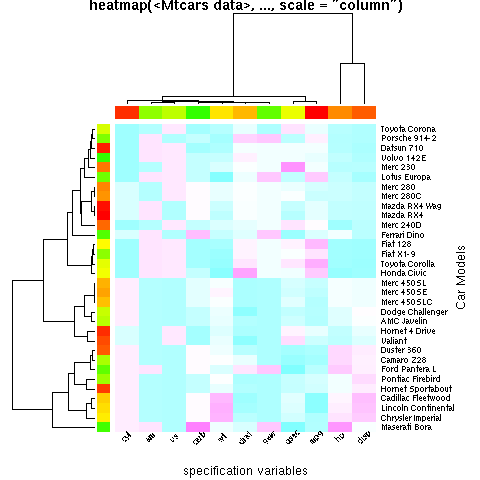
Najpierw przykład z ?heatmap
x <- as.matrix(mtcars)
rc <- rainbow(nrow(x), start = 0, end = .3)
cc <- rainbow(ncol(x), start = 0, end = .3)
hv <- heatmap(x, col = cm.colors(256), scale = "column",
RowSideColors = rc, ColSideColors = cc, margins = c(5,10),
xlab = "specification variables", ylab = "Car Models",
main = "heatmap(<Mtcars data>, ..., scale = \"column\")")
hv zawierają informacje potrzebne do zmiany kolejności colnames z mtcars w jego komponencie $colInd: {]}
> hv$colInd
[1] 2 9 8 11 6 5 10 7 1 4 3
Używasz tego tak, jakbyś miał wyjście z order np.:
> colnames(mtcars)[hv$colInd]
[1] "cyl" "am" "vs" "carb" "wt" "drat" "gear" "qsec" "mpg" "hp"
[11] "disp"
Teraz Użyj tego, aby wygenerować etykiety chcemy w odpowiedniej kolejności:
labs <- colnames(mtcars)[hv$colInd]
Następnie ponownie wywołujemy heatmap, ale tym razem określamy labCol = "", aby wyłączyć etykietowanie zmiennych kolumn(używając ciągów o zerowej długości). Używamy również wywołania text, aby narysować etykiety pod żądanym kątem. Wezwanie do text to:
text(x = seq_along(labs), y = -0.2, srt = 45, labels = labs, xpd = TRUE)
Co jest w zasadzie tym, co masz w swoim pytaniu. Graj z wartością y, ponieważ musisz dostosować ją do długości łańcuchów, aby etykiety nie pokrywały się z image fabuła Określamy labels = labs, aby przejść w etykietach, które chcemy narysować w wymaganej kolejności. Całe wywołanie text jest przekazywane do add.expr bez cytatu. Oto cała rozmowa:
hv <- heatmap(x, col = cm.colors(256), scale = "column",
RowSideColors = rc, ColSideColors = cc, margins = c(5,10),
xlab = "specification variables", ylab = "Car Models",
labCol = "",
main = "heatmap(<Mtcars data>, ..., scale = \"column\")",
add.expr = text(x = seq_along(labs), y = -0.2, srt = 45,
labels = labs, xpd = TRUE))
Co daje:
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2013-03-19 17:20:26
Szukam również metody obracania tekstu etykiety za pomocą heatmap. W końcu udało mi się znaleźć takie rozwiązanie:
library(gplots)
library(RColorBrewer)
heatmap.2(x,col=rev(brewer.pal(11,"Spectral")),cexRow=1,cexCol=1,margins=c(12,8),trace="none",srtCol=45)
Kluczowym argumentem jest srtCol(or srtRow for row labels), który jest używany do obracania etykiet kolumn w gplots.
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2013-11-14 14:17:09
Najnowsza wersja pheatmap (1.0.12) wersja z 2019-01-04 wspiera to argumentem angle_col.
#example data
d <- matrix(rnorm(25), 5, 5)
colnames(d) = paste("bip", 1:5, sep = "")
rownames(d) = paste("blob", 1:5, sep = "")
#update to latest version on CRAN
install.packages("pheatmap")
library("pheatmap")
pheatmap(d, angle_col = 45)
Stworzyłem pakiet na Githubie z ulepszoną wersją funkcji heatmap.2. Umożliwia To dostosowanie etykiet osi, w tym argumentu srtCol przekazywanego do funkcji axis. Można go zainstalować z: https://github.com/TomKellyGenetics/heatmap.2x
library("devtools")
install_github("TomKellyGenetics/heatmap.2x")
library("heatmap.2x")
heatmap.2x(d, scale = "none", trace = "none", col = heat.colors, srtCol = 45)
Od wersji 2.12.1 z gplots, funkcja heatmap.2 obsługuje również argument srtCol.
library("gplots")
heatmap.2(d, scale = "none", trace = "none", srtCol = 45)
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2019-02-28 06:50:03
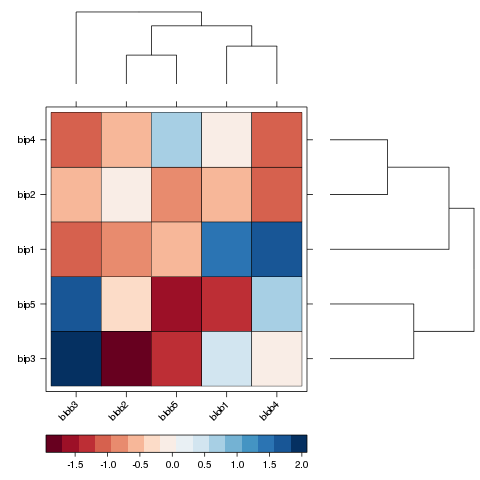
Rozwiązanie za pomocą lattice::levelplot i latticeExtra::dendrogramGrob:
library(lattice)
library(latticeExtra)
Przykładowe dane:
d <- matrix(rnorm(25), 5, 5)
colnames(d) = paste("bip", 1:5, sep = "")
rownames(d) = paste("blob", 1:5, sep = "")
Należy zdefiniować dendrogramy dla wierszy i kolumn (obliczone
wewnętrznie w heatmap):
dd.row <- as.dendrogram(hclust(dist(d)))
row.ord <- order.dendrogram(dd.row)
dd.col <- as.dendrogram(hclust(dist(t(d))))
col.ord <- order.dendrogram(dd.col)
I przekazać je do funkcji dendrogramGrob w legend
argument levelplot.
Zdefiniowałem nowy motyw z kolorami z RColorBrewer i
zmodyfikowano szerokość i kolor obramowań komórek za pomocą border
i border.lwd:
myTheme <- custom.theme(region=brewer.pal(n=11, 'RdBu'))
levelplot(d[row.ord, col.ord],
aspect = "fill", xlab='', ylab='',
scales = list(x = list(rot = 45)),
colorkey = list(space = "bottom"),
par.settings=myTheme,
border='black', border.lwd=.6,
legend =
list(right =
list(fun = dendrogramGrob,
args =
list(x = dd.col, ord = col.ord,
side = "right",
size = 10)),
top =
list(fun = dendrogramGrob,
args =
list(x = dd.row,
side = "top"))))
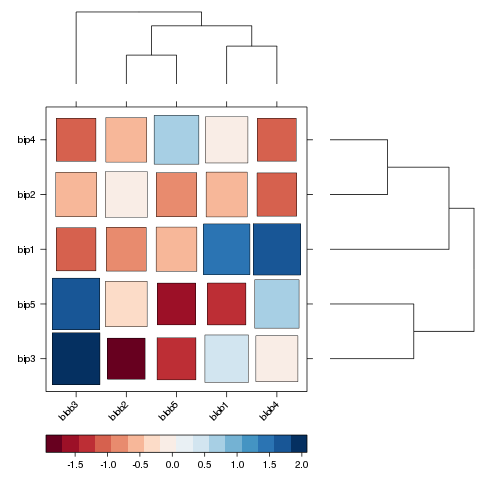
Możesz nawet użyć argumentu shrink, aby skaluj rozmiar komórek
proporcjonalna do ich wartości.
levelplot(d[row.ord, col.ord],
aspect = "fill", xlab='', ylab='',
scales = list(x = list(rot = 45)),
colorkey = list(space = "bottom"),
par.settings=myTheme,
border='black', border.lwd=.6,
shrink=c(.75, .95),
legend =
list(right =
list(fun = dendrogramGrob,
args =
list(x = dd.col, ord = col.ord,
side = "right",
size = 10)),
top =
list(fun = dendrogramGrob,
args =
list(x = dd.row,
side = "top"))))
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2013-03-20 22:22:42
Udało mi się wziąć odpowiedź Gavina Simpsona i przyciąć ją trochę do pracy dla mnie dla prostych celów prototypowania, gdzie data1 jest odczyt.obiekt csv () i data1_matrix oczywiście macierz wytworzona z tego
heatmap(data_matrix, Rowv=NA, Colv=NA, col=heat.colors(64), scale='column', margins=c(5,10),
labCol="", add.expr = text(x = seq_along(colnames(data1)), y=-0.2, srt=45,
labels=colnames(data1), xpd=TRUE))
Kluczowym bitem do tego działania jest część przed add.expr bitem, w którym ustawił labCol na"", co jest konieczne, aby zapobiec nakładaniu się pierwszych (prostych) etykiet na nowe 45 stopni
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2013-04-07 01:59:09



