Dla każdego wiersza w ramce danych R
Mam dataframe i dla każdego wiersza w tym dataframe muszę wykonać kilka skomplikowanych poszukiwań i dołączyć niektóre dane do pliku.
Ramka danych zawiera wyniki naukowe dla wybranych studni z 96 płyt studni wykorzystywanych w badaniach biologicznych, więc chcę zrobić coś takiego:
for (well in dataFrame) {
wellName <- well$name # string like "H1"
plateName <- well$plate # string like "plate67"
wellID <- getWellID(wellName, plateName)
cat(paste(wellID, well$value1, well$value2, sep=","), file=outputFile)
}
W moim proceduralnym świecie, zrobiłbym coś takiego:
for (row in dataFrame) {
#look up stuff using data from the row
#write stuff to the file
}
Jak to zrobić?
8 answers
Możesz tego spróbować, używając apply() function
> d
name plate value1 value2
1 A P1 1 100
2 B P2 2 200
3 C P3 3 300
> f <- function(x, output) {
wellName <- x[1]
plateName <- x[2]
wellID <- 1
print(paste(wellID, x[3], x[4], sep=","))
cat(paste(wellID, x[3], x[4], sep=","), file= output, append = T, fill = T)
}
> apply(d, 1, f, output = 'outputfile')
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2014-01-15 00:26:56
Możesz użyć by() Funkcja:
by(dataFrame, 1:nrow(dataFrame), function(row) dostuff)
Ale iteracja wierszy bezpośrednio w ten sposób rzadko jest tym, czego chcesz; powinieneś zamiast tego spróbować wektoryzować. Czy mogę zapytać, co robi rzeczywista praca w pętli?
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2014-01-15 00:26:32
Po pierwsze, punkt Jonathana o wektoryzacji jest prawidłowy. Jeśli funkcja getWellID() jest wektoryzowana, możesz pominąć pętlę i po prostu użyć cat lub write.csv:
write.csv(data.frame(wellid=getWellID(well$name, well$plate),
value1=well$value1, value2=well$value2), file=outputFile)
Jeśli getWellID () nie jest wektoryzowane, to zalecenie Jonathana dotyczące używania by lub sugestia knguyena dotycząca apply powinny zadziałać.
W przeciwnym razie, jeśli naprawdę chcesz użyć for, możesz zrobić coś takiego:
for(i in 1:nrow(dataFrame)) {
row <- dataFrame[i,]
# do stuff with row
}
Możesz również spróbować użyć pakietu foreach, chociaż wymaga to od Ciebie zna tę składnię. Oto prosty przykład:
library(foreach)
d <- data.frame(x=1:10, y=rnorm(10))
s <- foreach(d=iter(d, by='row'), .combine=rbind) %dopar% d
Ostatnią opcją jest użycie funkcji z pakietu plyr, w którym to przypadku konwencja będzie bardzo podobna do funkcji apply.
library(plyr)
ddply(dataFrame, .(x), function(x) { # do stuff })
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2009-11-09 14:58:07
Używam tej prostej funkcji użytkowej:
rows = function(tab) lapply(
seq_len(nrow(tab)),
function(i) unclass(tab[i,,drop=F])
)
Lub szybsza, mniej przejrzysta forma:
rows = function(x) lapply(seq_len(nrow(x)), function(i) lapply(x,"[",i))
Ta funkcja po prostu dzieli dane.ramka do listy wierszy. Następnie możesz zrobić normalne " dla " na tej liście:
tab = data.frame(x = 1:3, y=2:4, z=3:5)
for (A in rows(tab)) {
print(A$x + A$y * A$z)
}
Twój kod z pytania będzie działał z minimalną modyfikacją:
for (well in rows(dataFrame)) {
wellName <- well$name # string like "H1"
plateName <- well$plate # string like "plate67"
wellID <- getWellID(wellName, plateName)
cat(paste(wellID, well$value1, well$value2, sep=","), file=outputFile)
}
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2017-04-12 19:33:30
Byłem ciekaw czasu wykonania opcji bez wektoryzowanych. W tym celu użyłem funkcji f zdefiniowanej przez knguyen
f <- function(x, output) {
wellName <- x[1]
plateName <- x[2]
wellID <- 1
print(paste(wellID, x[3], x[4], sep=","))
cat(paste(wellID, x[3], x[4], sep=","), file= output, append = T, fill = T)
}
I ramka danych, taka jak w jego przykładzie:
n = 100; #number of rows for the data frame
d <- data.frame( name = LETTERS[ sample.int( 25, n, replace=T ) ],
plate = paste0( "P", 1:n ),
value1 = 1:n,
value2 = (1:n)*10 )
Dodałem dwie wektoryzowane funkcje (na pewno szybsze niż pozostałe), aby porównać podejście cat() z zapisem.tabela() 1...
library("ggplot2")
library( "microbenchmark" )
library( foreach )
library( iterators )
tm <- microbenchmark(S1 =
apply(d, 1, f, output = 'outputfile1'),
S2 =
for(i in 1:nrow(d)) {
row <- d[i,]
# do stuff with row
f(row, 'outputfile2')
},
S3 =
foreach(d1=iter(d, by='row'), .combine=rbind) %dopar% f(d1,"outputfile3"),
S4= {
print( paste(wellID=rep(1,n), d[,3], d[,4], sep=",") )
cat( paste(wellID=rep(1,n), d[,3], d[,4], sep=","), file= 'outputfile4', sep='\n',append=T, fill = F)
},
S5 = {
print( (paste(wellID=rep(1,n), d[,3], d[,4], sep=",")) )
write.table(data.frame(rep(1,n), d[,3], d[,4]), file='outputfile5', row.names=F, col.names=F, sep=",", append=T )
},
times=100L)
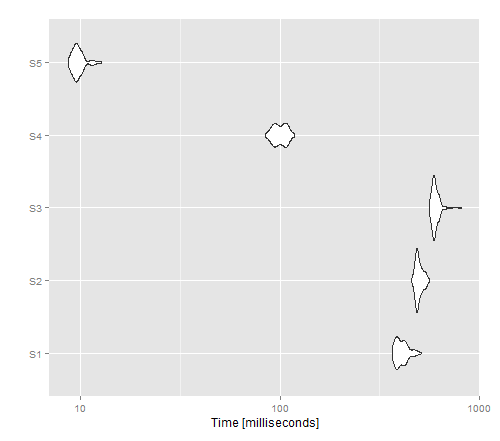
autoplot(tm)
Wynikowy obraz pokazuje, że apply daje najlepszą wydajność dla wersji bez wektoryzowanej, podczas gdy write.tabela() wydaje się przewyższać cat ().
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2015-07-14 14:13:43
Myślę, że najlepszym sposobem, aby to zrobić z podstawowym R jest:
for( i in rownames(df) )
print(df[i, "column1"])
Przewaga nad for(I in 1:nrow(df))-podejście polega na tym, że nie wpadasz w kłopoty, jeśli DF jest pusty i nrow (df)=0.
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2017-07-16 16:07:39
Możesz użyć funkcji by_row z pakietu purrrlyr w tym celu:
myfn <- function(row) {
#row is a tibble with one row, and the same
#number of columns as the original df
#If you'd rather it be a list, you can use as.list(row)
}
purrrlyr::by_row(df, myfn)
Domyślnie zwracana wartość z myfn jest umieszczana w nowej kolumnie listy W df o nazwie .out.
Jeśli jest to jedyne wyjście, którego pragniesz, możesz napisać purrrlyr::by_row(df, myfn)$.out
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2017-06-03 19:26:01
Cóż, skoro prosiłeś o odpowiedniki R w innych językach, próbowałem to zrobić. Wygląda na to, że działa, chociaż nie przyjrzałem się, która technika jest bardziej efektywna w R.
> myDf <- head(iris)
> myDf
Sepal.Length Sepal.Width Petal.Length Petal.Width Species
1 5.1 3.5 1.4 0.2 setosa
2 4.9 3.0 1.4 0.2 setosa
3 4.7 3.2 1.3 0.2 setosa
4 4.6 3.1 1.5 0.2 setosa
5 5.0 3.6 1.4 0.2 setosa
6 5.4 3.9 1.7 0.4 setosa
> nRowsDf <- nrow(myDf)
> for(i in 1:nRowsDf){
+ print(myDf[i,4])
+ }
[1] 0.2
[1] 0.2
[1] 0.2
[1] 0.2
[1] 0.2
[1] 0.4
Dla kolumn kategorycznych, pobierze Ci ramkę danych, którą możesz wpisać używając jako.znak () w razie potrzeby.
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2015-02-13 15:04:03