Generowanie mapy ciepła w MatPlotLib za pomocą zestawu danych rozproszonych
Mam zestaw punktów danych X,Y (około 10k), które są łatwe do wykreślenia jako wykres punktowy, ale które chciałbym przedstawić jako heatmap.
Przejrzałem przykłady w MatPlotLib i wszystkie wydają się już zaczynać od wartości komórek heatmap, aby wygenerować obraz.
Czy istnieje metoda, która konwertuje kilka x, Y, wszystkie różne, do heatmap (gdzie strefy o wyższej częstotliwości x, y byłyby "cieplejsze")?
8 answers
Jeśli nie chcesz heksagonów, możesz użyć funkcji numpy histogram2d:
import numpy as np
import numpy.random
import matplotlib.pyplot as plt
# Generate some test data
x = np.random.randn(8873)
y = np.random.randn(8873)
heatmap, xedges, yedges = np.histogram2d(x, y, bins=50)
extent = [xedges[0], xedges[-1], yedges[0], yedges[-1]]
plt.clf()
plt.imshow(heatmap.T, extent=extent, origin='lower')
plt.show()
To sprawia, że mapa cieplna 50x50. Jeśli chcesz, powiedzmy, 512x384, możesz umieścić bins=(512, 384) w wywołaniu do histogram2d.
Przykład: 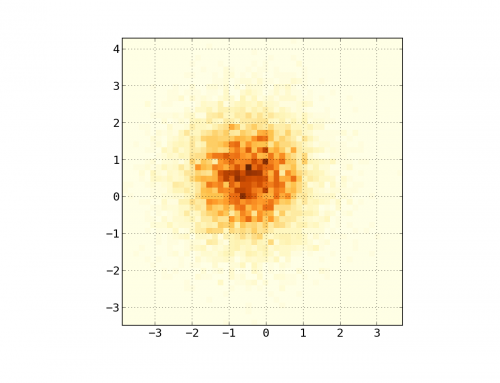
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2016-10-27 23:21:26
W Matplotlib leksykon, myślę, że chceszhexbin fabuła.
Jeśli nie jesteś zaznajomiony z tego typu wykresem, jest to po prostu biwarialny histogram , w którym płaszczyzna xy jest tessellowana przez regularną siatkę sześciokątów.
Więc z histogramu można po prostu policzyć liczbę punktów przypadających na każdy sześciokąt, dyskrecjonować obszar wykresu jako zbiór okien , przypisać każdy punkt do jednego z tych okien; na koniec zmapować okna na kolor array , i masz diagram heksabiny.
Choć rzadziej używane niż np. okręgi lub kwadraty, to heksagony są lepszym wyborem dla geometrii pojemnika binning jest intuicyjne:
-
Sześciokąty mają symetrię najbliższego sąsiada (np. kwadraty nie, np. odległość od punktu na granicy kwadratu do punktu wewnątrz tego kwadratu nie wszędzie jest równe) i
Sześciokąt jest najwyższym N-wielokątem, który daje płaszczyznę regularną teselacja (tzn. możesz bezpiecznie wymodelować podłogę kuchenną za pomocą sześciokątnych płytek, ponieważ nie będziesz miał żadnej pustej przestrzeni między płytkami po zakończeniu-nie dotyczy to wszystkich innych wyższych-n, N >= 7, wielokątów).
(Matplotlib używa terminu hexbin plot; tak jak (AFAIK) wszystkie biblioteki plotowania dla R ; nadal Nie wiem, czy jest to ogólnie przyjęte określenie dla działek tego typu, chociaż podejrzewa się, że jest prawdopodobne, biorąc pod uwagę, że hexbin jest skrótem od hexagonal binning, który opisuje istotny krok w przygotowaniu danych do wyświetlenia.)
from matplotlib import pyplot as PLT
from matplotlib import cm as CM
from matplotlib import mlab as ML
import numpy as NP
n = 1e5
x = y = NP.linspace(-5, 5, 100)
X, Y = NP.meshgrid(x, y)
Z1 = ML.bivariate_normal(X, Y, 2, 2, 0, 0)
Z2 = ML.bivariate_normal(X, Y, 4, 1, 1, 1)
ZD = Z2 - Z1
x = X.ravel()
y = Y.ravel()
z = ZD.ravel()
gridsize=30
PLT.subplot(111)
# if 'bins=None', then color of each hexagon corresponds directly to its count
# 'C' is optional--it maps values to x-y coordinates; if 'C' is None (default) then
# the result is a pure 2D histogram
PLT.hexbin(x, y, C=z, gridsize=gridsize, cmap=CM.jet, bins=None)
PLT.axis([x.min(), x.max(), y.min(), y.max()])
cb = PLT.colorbar()
cb.set_label('mean value')
PLT.show()
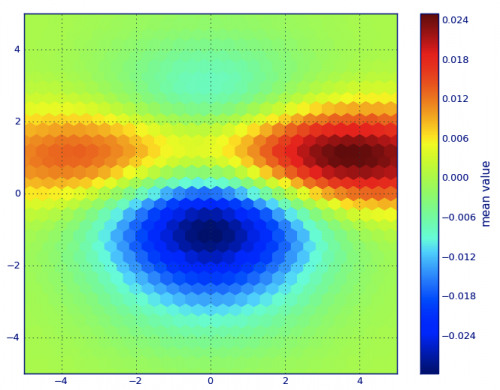
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2014-04-24 02:51:05
Zamiast używać np.hist2d, który ogólnie produkuje dość brzydkie histogramy, chciałbym ponownie wykorzystać py-sphviewer , pakiet Pythona do renderowania symulacji cząstek przy użyciu adaptacyjnego jądra wygładzającego i który można łatwo zainstalować z pip (patrz dokumentacja strony internetowej). Rozważ następujący kod, który jest oparty na przykładzie:
import numpy as np
import numpy.random
import matplotlib.pyplot as plt
import sphviewer as sph
def myplot(x, y, nb=32, xsize=500, ysize=500):
xmin = np.min(x)
xmax = np.max(x)
ymin = np.min(y)
ymax = np.max(y)
x0 = (xmin+xmax)/2.
y0 = (ymin+ymax)/2.
pos = np.zeros([3, len(x)])
pos[0,:] = x
pos[1,:] = y
w = np.ones(len(x))
P = sph.Particles(pos, w, nb=nb)
S = sph.Scene(P)
S.update_camera(r='infinity', x=x0, y=y0, z=0,
xsize=xsize, ysize=ysize)
R = sph.Render(S)
R.set_logscale()
img = R.get_image()
extent = R.get_extent()
for i, j in zip(xrange(4), [x0,x0,y0,y0]):
extent[i] += j
print extent
return img, extent
fig = plt.figure(1, figsize=(10,10))
ax1 = fig.add_subplot(221)
ax2 = fig.add_subplot(222)
ax3 = fig.add_subplot(223)
ax4 = fig.add_subplot(224)
# Generate some test data
x = np.random.randn(1000)
y = np.random.randn(1000)
#Plotting a regular scatter plot
ax1.plot(x,y,'k.', markersize=5)
ax1.set_xlim(-3,3)
ax1.set_ylim(-3,3)
heatmap_16, extent_16 = myplot(x,y, nb=16)
heatmap_32, extent_32 = myplot(x,y, nb=32)
heatmap_64, extent_64 = myplot(x,y, nb=64)
ax2.imshow(heatmap_16, extent=extent_16, origin='lower', aspect='auto')
ax2.set_title("Smoothing over 16 neighbors")
ax3.imshow(heatmap_32, extent=extent_32, origin='lower', aspect='auto')
ax3.set_title("Smoothing over 32 neighbors")
#Make the heatmap using a smoothing over 64 neighbors
ax4.imshow(heatmap_64, extent=extent_64, origin='lower', aspect='auto')
ax4.set_title("Smoothing over 64 neighbors")
plt.show()
, który tworzy następujący obrazek:
Jak widzisz, zdjęcia wyglądają całkiem ładnie, a my jesteśmy w stanie aby zidentyfikować na niej różne podstruktury. Obrazy te są konstruowane z rozłożeniem danej wagi dla każdego punktu w określonej domenie, określonej przez długość wygładzania, która z kolei jest określona przez odległość do bliższego nb sąsiada (na przykład wybrałem 16, 32 i 64). Tak więc regiony o wyższej gęstości zazwyczaj są rozłożone na mniejsze regiony w porównaniu do regionów o niższej gęstości.
Funkcja myplot jest po prostu bardzo prostą funkcją, którą napisałem, aby dać dane x, y do py-sphviewer zrobić magię.
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2016-04-09 10:06:31
Jeśli używasz 1.2.x
x = randn(100000) y = randn(100000) hist2d(x,y,bins=100);
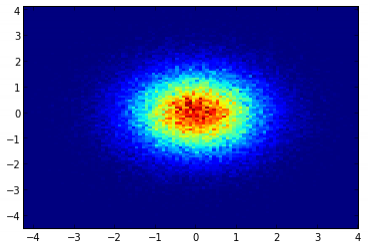
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2012-11-05 14:19:29
Edit: aby uzyskać lepsze przybliżenie odpowiedzi Alejandro, zobacz poniżej.
Wiem, że to stare pytanie, ale chciałem dodać coś do anwsera Alejandro: jeśli chcesz mieć ładny wygładzony obraz bez używania py-sphviewer, możesz zamiast tego użyć np.histogram2d i zastosować filtr Gaussa (z scipy.ndimage.filters) do heatmapy:
import numpy as np
import matplotlib.pyplot as plt
import matplotlib.cm as cm
from scipy.ndimage.filters import gaussian_filter
def myplot(x, y, s, bins=1000):
heatmap, xedges, yedges = np.histogram2d(x, y, bins=bins)
heatmap = gaussian_filter(heatmap, sigma=s)
extent = [xedges[0], xedges[-1], yedges[0], yedges[-1]]
return heatmap.T, extent
fig, axs = plt.subplots(2, 2)
# Generate some test data
x = np.random.randn(1000)
y = np.random.randn(1000)
sigmas = [0, 16, 32, 64]
for ax, s in zip(axs.flatten(), sigmas):
if s == 0:
ax.plot(x, y, 'k.', markersize=5)
ax.set_title("Scatter plot")
else:
img, extent = myplot(x, y, s)
ax.imshow(img, extent=extent, origin='lower', cmap=cm.jet)
ax.set_title("Smoothing with $\sigma$ = %d" % s)
plt.show()
Produkuje:
Wykres punktowy i s=16 wykreślone na wierzchu dla Agape Gal ' lo (kliknij Dla Lepszego Widok):
Jedną z różnic zauważyłem z moim podejściem filtra Gaussa i podejście Alejandro było to, że jego metoda pokazuje lokalne struktury znacznie lepiej niż moja. Dlatego zaimplementowałem prostą metodę najbliższego sąsiada na poziomie pikseli. Metoda ta oblicza dla każdego piksela odwrotną sumę odległości n najbliższych punktów w danych. Ta metoda jest w wysokiej rozdzielczości dość kosztowna obliczeniowo i myślę, że jest szybszy sposób, więc daj mi znać, jeśli masz jakieś ulepszenia. W każdym razie, oto kod:
import numpy as np
import matplotlib.pyplot as plt
import matplotlib.cm as cm
def data_coord2view_coord(p, vlen, pmin, pmax):
dp = pmax - pmin
dv = (p - pmin) / dp * vlen
return dv
def nearest_neighbours(xs, ys, reso, n_neighbours):
im = np.zeros([reso, reso])
extent = [np.min(xs), np.max(xs), np.min(ys), np.max(ys)]
xv = data_coord2view_coord(xs, reso, extent[0], extent[1])
yv = data_coord2view_coord(ys, reso, extent[2], extent[3])
for x in range(reso):
for y in range(reso):
xp = (xv - x)
yp = (yv - y)
d = np.sqrt(xp**2 + yp**2)
im[y][x] = 1 / np.sum(d[np.argpartition(d.ravel(), n_neighbours)[:n_neighbours]])
return im, extent
n = 1000
xs = np.random.randn(n)
ys = np.random.randn(n)
resolution = 250
fig, axes = plt.subplots(2, 2)
for ax, neighbours in zip(axes.flatten(), [0, 16, 32, 64]):
if neighbours == 0:
ax.plot(xs, ys, 'k.', markersize=2)
ax.set_aspect('equal')
ax.set_title("Scatter Plot")
else:
im, extent = nearest_neighbours(xs, ys, resolution, neighbours)
ax.imshow(im, origin='lower', extent=extent, cmap=cm.jet)
ax.set_title("Smoothing over %d neighbours" % neighbours)
ax.set_xlim(extent[0], extent[1])
ax.set_ylim(extent[2], extent[3])
plt.show()
Wynik:
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2018-09-11 08:06:27
Seaborn ma teraz funkcję jointplot , która powinna działać ładnie tutaj:
import numpy as np
import seaborn as sns
import matplotlib.pyplot as plt
# Generate some test data
x = np.random.randn(8873)
y = np.random.randn(8873)
sns.jointplot(x=x, y=y, kind='hex')
plt.show()
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2017-03-02 23:57:57
Stwórz 2-wymiarową tablicę, która odpowiada komórkom na ostatecznym obrazie, nazywaną say heatmap_cells i utwórz ją jako instancję wszystkich zer.
Wybierz dwa czynniki skalujące, które definiują różnicę między każdym elementem tablicy w jednostkach rzeczywistych, dla każdego wymiaru, powiedzmy x_scale i y_scale. Wybierz takie, aby wszystkie twoje punkty danych mieściły się w granicach tablicy heatmap.
Dla każdego surowego punktu danych z x_value i y_value:
heatmap_cells[floor(x_value/x_scale),floor(y_value/y_scale)]+=1
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2010-03-03 12:37:50
Pierwsze pytanie brzmiało:.. jak przekonwertować wartości rozproszone na wartości siatki, prawda?
histogram2d liczy częstotliwość na komórkę, jednak jeśli masz inne dane na komórkę niż tylko częstotliwość, potrzebujesz dodatkowej pracy.
x = data_x # between -10 and 4, log-gamma of an svc
y = data_y # between -4 and 11, log-C of an svc
z = data_z #between 0 and 0.78, f1-values from a difficult dataset
Więc mam zbiór danych z wynikami z dla współrzędnych X i Y. Obliczałem jednak kilka punktów poza obszarem zainteresowania (duże luki) i sterty punktów w małym obszarze zainteresowania.
Tak tutaj staje się trudniejsze, ale i więcej zabawy. Niektóre biblioteki (sorry):
from matplotlib import pyplot as plt
from matplotlib import cm
import numpy as np
from scipy.interpolate import griddata
Pyplot to mój silnik graficzny, cm to gama kolorowych map z pewnym initeresting wybór. numpy do obliczeń, i griddata do dołączania wartości do stałej siatki.
Ostatni jest ważny, zwłaszcza, że częstotliwość punktów xy nie jest równomiernie rozłożona w moich danych. Po pierwsze, zacznijmy od pewnych granic dopasowanych do moich danych i dowolnego rozmiaru siatki. Oryginalnych danych posiada punkty danych również poza tymi x i / align = "left" /
#determine grid boundaries
gridsize = 500
x_min = -8
x_max = 2.5
y_min = -2
y_max = 7
Zdefiniowaliśmy więc siatkę o wielkości 500 pikseli pomiędzy wartościami min i max X i y.
W moich danych jest o wiele więcej niż 500 wartości dostępnych w obszarze o dużym zainteresowaniu; podczas gdy w obszarze o niskim zainteresowaniu nie ma nawet 200 wartości w całkowitej siatce; między graficznymi granicami x_min i x_max jest jeszcze mniej.
Więc dla uzyskania ładnego obrazu, zadaniem jest uzyskanie średniej dla wysokich wartości zainteresowania i wypełnienie luk gdzie indziej.
Definiuję teraz swoją siatkę. Dla każdej pary xx-yy chcę mieć kolor.
xx = np.linspace(x_min, x_max, gridsize) # array of x values
yy = np.linspace(y_min, y_max, gridsize) # array of y values
grid = np.array(np.meshgrid(xx, yy.T))
grid = grid.reshape(2, grid.shape[1]*grid.shape[2]).T
Griddata oblicza jedną wartość na punkt w siatce, za pomocą predefiniowanej metody. Wybieram "najbliższy" - puste punkty siatki zostaną wypełnione wartościami od najbliższego sąsiada. Wygląda to tak, jakby obszary o mniejszej ilości informacji miały większe komórki(nawet jeśli tak nie jest). Można wybrać interpolację "liniowe", wtedy obszary z mniejszą ilością informacji wyglądają mniej ostro. Kwestia gustu.
points = np.array([x, y]).T # because griddata wants it that way
z_grid2 = griddata(points, z, grid, method='nearest')
# you get a 1D vector as result. Reshape to picture format!
z_grid2 = z_grid2.reshape(xx.shape[0], yy.shape[0])
fig = plt.figure(1, figsize=(10, 10))
ax1 = fig.add_subplot(111)
ax1.imshow(z_grid2, extent=[x_min, x_max,y_min, y_max, ],
origin='lower', cmap=cm.magma)
ax1.set_title("SVC: empty spots filled by nearest neighbours")
ax1.set_xlabel('log gamma')
ax1.set_ylabel('log C')
plt.show()
Wokół spiczastej Części W Kształcie Litery V, widzisz, wykonałem wiele obliczeń podczas moich poszukiwań sweet spot, podczas gdy mniej interesujące części prawie wszędzie mają niższą rozdzielczość.
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2018-07-17 14:00:48





