pymc3: wiele obserwowanych wartości
Mam kilka danych obserwacyjnych, dla których chciałbym oszacować parametry, i pomyślałem, że będzie to dobra okazja, aby wypróbować PYMC3.
Moje dane są uporządkowane jako seria rekordów. Każdy zapis zawiera parę obserwacji, które odnoszą się do ustalonego okresu JEDNEJ godziny. Jedna obserwacja to całkowita liczba zdarzeń, które wystąpiły w ciągu danej godziny. Inną obserwacją jest liczba sukcesów w tym okresie. Tak więc, na przykład, punkt danych można określić, że w danym okresie 1 godziny, było 1000 zdarzeń w sumie, a z tych 1000, 100 były sukcesy. W innym okresie może być łącznie 1000000 wydarzeń, z czego 120000 to sukcesy. Wariancja obserwacji nie jest stała i zależy od całkowitej liczby zdarzeń, i to częściowo ten efekt, który chciałbym kontrolować i modelować.
Mój pierwszy krok w tym, aby oszacować podstawowy wskaźnik sukcesu. Przygotowałem poniższy kod, który ma na celu naśladowanie sytuacji poprzez dostarczenie dwóch zestawów "obserwowanych" danych, generując je za pomocą scipy. Jednak nie działa prawidłowo.
Spodziewam się, że znajdzie:
- loss_lambda_factor jest w przybliżeniu 0.1
- total_lambda (i total_lambda_mu) wynosi około 120.
Zamiast tego model zbiega się bardzo szybko, ale na nieoczekiwaną odpowiedź.
- total_lambda i total_lambda_mu są odpowiednio ostrymi szczytami wokół 5e5.
- loss_lambda_factor jest w przybliżeniu równy 0.
Traceplot (którego nie mogę opublikować ze względu na reputację poniżej 10) jest dość nieciekawy - szybka zbieżność i ostre szczyty w liczbach, które nie odpowiadają danym wejściowym. Jestem ciekaw, czy w podejściu, które stosuję, jest coś zasadniczo złego. Jak należy zmodyfikować poniższy kod, aby dać poprawny / oczekiwany wynik?
from pymc import Model, Uniform, Normal, Poisson, Metropolis, traceplot
from pymc import sample
import scipy.stats
totalRates = scipy.stats.norm(loc=120, scale=20).rvs(size=10000)
totalCounts = scipy.stats.poisson.rvs(mu=totalRates)
successRate = 0.1*totalRates
successCounts = scipy.stats.poisson.rvs(mu=successRate)
with Model() as success_model:
total_lambda_tau= Uniform('total_lambda_tau', lower=0, upper=100000)
total_lambda_mu = Uniform('total_lambda_mu', lower=0, upper=1000000)
total_lambda = Normal('total_lambda', mu=total_lambda_mu, tau=total_lambda_tau)
total = Poisson('total', mu=total_lambda, observed=totalCounts)
loss_lambda_factor = Uniform('loss_lambda_factor', lower=0, upper=1)
success_rate = Poisson('success_rate', mu=total_lambda*loss_lambda_factor, observed=successCounts)
with success_model:
step = Metropolis()
success_samples = sample(20000, step) #, start)
plt.figure(figsize=(10, 10))
_ = traceplot(success_samples)
2 answers
Nie ma nic zasadniczo złego w twoim podejściu, z wyjątkiem pułapek każdej bayesowskiej analizy MCMC: (1) non-convergence, (2) priors, (3) model.
Non-convergence : znajduję traceplot, który wygląda tak:
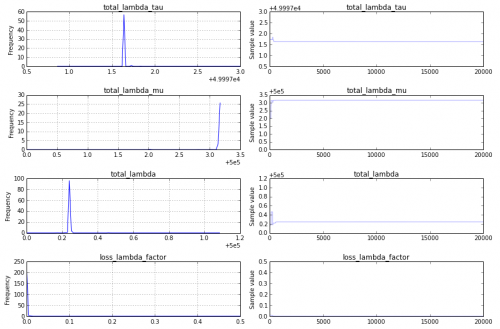
To nie jest dobra rzecz, i aby lepiej zrozumieć dlaczego, zmieniłbym kod traceplot, aby pokazać tylko drugą połowę śladu, traceplot(success_samples[10000:]):
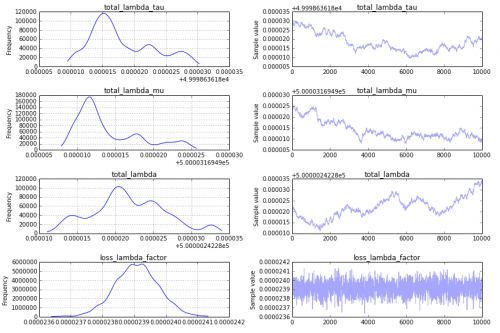
Przeor : jedno wielkie wyzwanie dla zbieżności jest Twój przeor na total_lambda_tau, który jest przykładem pułapki w modelowaniu Bayesowskim. Chociaż użycie wcześniejszego Uniform('total_lambda_tau', lower=0, upper=100000) może wydawać się dość nieinformatyczne, efekt tego polega na tym, że jesteś całkiem pewien, że total_lambda_tau jest duży. Na przykład prawdopodobieństwo, że jest mniejsza niż 10 jest .0001. Zmiana przed
total_lambda_tau= Uniform('total_lambda_tau', lower=0, upper=100)
total_lambda_mu = Uniform('total_lambda_mu', lower=0, upper=1000)
Powoduje, że traceplot jest bardziej obiecujący:
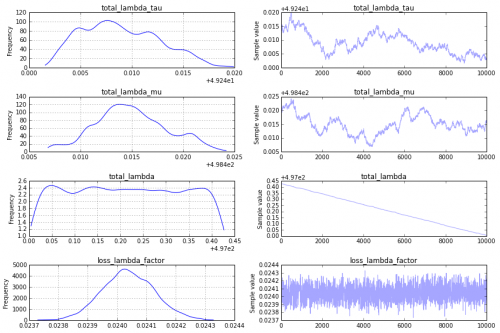
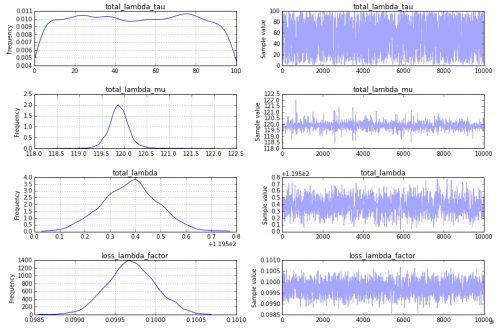
To nadal nie jest to, czego szukam w traceplot, jednak, aby uzyskać coś bardziej zadowalające, proponuję użyć kroku "sequential scan Metropolis" (czyli tego, co domyślnie PyMC2 dla analogicznego modelu). Można to określić w następujący sposób:
step = pm.CompoundStep([pm.Metropolis([total_lambda_mu]),
pm.Metropolis([total_lambda_tau]),
pm.Metropolis([total_lambda]),
pm.Metropolis([loss_lambda_factor]),
])
To tworzy traceplot, który wydaje się akceptowalny:
model: Jak odpowiedział @KaiLondenberg, podejście, które przyjąłeś z notami na total_lambda_tau i total_lambda_mu, nie jest standardowym podejściem. Opisujesz bardzo różne sumy zdarzeń (1000 w jednej godzinie i 1000 000 w następnej), ale twoje model zakłada, że będzie on normalnie dystrybuowany. W epidemiologii przestrzennej podejście, które widziałem dla analogicznych danych, jest modelem bardziej podobnym do tego:
import pymc as pm, theano.tensor as T
with Model() as success_model:
loss_lambda_rate = pm.Flat('loss_lambda_rate')
error = Poisson('error', mu=totalCounts*T.exp(loss_lambda_rate),
observed=successCounts)
Jestem pewien, że są inne sposoby, które będą wydawać się bardziej znane w innych społecznościach badawczych, jak również.
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2014-06-17 19:21:30
Widzę kilka potencjalnych problemów z modelem.
1.) Myślę, że liczy się sukces (tzw. błąd ?) powinien podążać za rozkładem Dwumianowym(N=całkowity,p=loss_lambda_factor), a nie Poissonem.
2.) Od czego zaczyna się łańcuch ? Zaczynając od konfiguracji MAP lub mle miałoby sens, chyba że użyjesz czystego próbkowania Gibbsa. W przeciwnym razie spalenie łańcucha może zająć dużo czasu, co może być tym, co się tutaj dzieje.
3.) Twój wybór hierarchicznego przeora dla total_lambda (tzn. normalna z dwoma jednolitymi wartościami na tych parametrach) zapewnia, że zbieżność łańcucha zajmie dużo czasu, chyba że mądrze wybierzesz swój start (jak w punkcie 2.). Zasadniczo wprowadzasz wiele niezauważalnych stopni swobody, aby łańcuch MCMC mógł się zgubić. Biorąc pod uwagę, że total_lambda musi być nonngative, wybrałbym jednorodny prior dla total_lambda w odpowiednim zakresie (na przykład od 0 do obserwowanego maksimum).
4.) Używasz samplera Metropolis. 20000 próbki mogą nie wystarczyć. Spróbuj 60000 i wyrzuć pierwsze 20000 jako wypalanie. Sampler Metropolis może trochę potrwać, aby dostroić rozmiar kroku, więc może być tak, że spędził pierwsze 20000 próbek, aby głównie odrzucić propozycje i dostroić. Spróbuj innych samplerów, takich jak NUTS.
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2014-06-17 08:07:27