dplyr na danych.table, czy naprawdę używam danych.stolik?
Jeśli używam składni dplyrna szczycie datatable, czy otrzymam wszystkie korzyści związane z szybkością datatable podczas korzystania ze składni dplyr? Innymi słowy, czy źle używam datatable, jeśli odpytywam go za pomocą składni dplyr? Czy też muszę użyć składni Pure datatable, aby wykorzystać całą jego moc?
Z góry dziękuję za wszelkie rady. Przykład Kodu:library(data.table)
library(dplyr)
diamondsDT <- data.table(ggplot2::diamonds)
setkey(diamondsDT, cut)
diamondsDT %>%
filter(cut != "Fair") %>%
group_by(cut) %>%
summarize(AvgPrice = mean(price),
MedianPrice = as.numeric(median(price)),
Count = n()) %>%
arrange(desc(Count))
Wyniki:
# cut AvgPrice MedianPrice Count
# 1 Ideal 3457.542 1810.0 21551
# 2 Premium 4584.258 3185.0 13791
# 3 Very Good 3981.760 2648.0 12082
# 4 Good 3928.864 3050.5 4906
Oto równoważność danych, którą wymyśliłem. Nie wiem, czy jest zgodny z DT good praktyka. Ale zastanawiam się, czy kod jest naprawdę bardziej wydajny niż składnia dplyr za sceną:
diamondsDT [cut != "Fair"
] [, .(AvgPrice = mean(price),
MedianPrice = as.numeric(median(price)),
Count = .N), by=cut
] [ order(-Count) ]
3 answers
Nie ma prostej/prostej odpowiedzi, ponieważ filozofie obu tych pakietów różnią się w pewnych aspektach. Więc niektóre kompromisy są nieuniknione. Oto niektóre z obaw, które mogą być konieczne do rozwiązania/rozważenia.
Operacje polegające na i (== filter() i slice() w dplyr)
Załóżmy {[7] } z powiedzmy 10 kolumnami. Rozważ te dane.wyrażenia tabeli:
DT[a > 1, .N] ## --- (1)
DT[a > 1, mean(b), by=.(c, d)] ## --- (2)
(1) podaje liczbę wierszy w DT, gdzie kolumna a > 1. (2) zwraca mean(b) zgrupowane według c,d dla tego samego wyrażenia w i Jak (1).
Powszechnie używane dplyr wyrażenia to:
DT %>% filter(a > 1) %>% summarise(n()) ## --- (3)
DT %>% filter(a > 1) %>% group_by(c, d) %>% summarise(mean(b)) ## --- (4)
Wyraźnie, data.kody tabel są krótsze. Ponadto są one również bardziej wydajne pamięci1. Dlaczego? Ponieważ w obu (3) i (4) filter() zwraca wiersze dla wszystkich 10 kolumn najpierw, gdy w (3) potrzebujemy tylko liczby wierszy, a w (4) potrzebujemy tylko kolumn b, c, d dla kolejnych operacji. Aby to przezwyciężyć, musimy select() kolumny apriori:
DT %>% select(a) %>% filter(a > 1) %>% summarise(n()) ## --- (5)
DT %>% select(a,b,c,d) %>% filter(a > 1) %>% group_by(c,d) %>% summarise(mean(b)) ## --- (6)
[[32]}Ważne jest, aby podkreślić główną filozoficzną różnicę między tymi dwoma pakietami: [33]}
W
data.tablelubimy trzymać te powiązane operacje razem, co pozwala spojrzeć naj-expression(z tego samego wywołania funkcji) i uświadomić sobie, że nie ma potrzeby stosowania żadnych kolumn w (1). Wyrażenie wijest obliczane, a.Njest tylko sumą tego wektora logicznego, który daje liczbę wierszy; cały podzbiór nigdy nie jest spełniony. W (2), tylko kolumny {[21] } są zmaterializowane w podzbiorze, Pozostałe Kolumny są ignorowane.Ale w
dplyr, filozofia jest mieć funkcję zrobić dokładnie jedną rzecz dobrze . Nie ma (przynajmniej obecnie) sposobu, aby stwierdzić, czy operacja pofilter()potrzebuje wszystkich filtrowanych kolumn. Musisz myśleć z wyprzedzeniem, jeśli chcesz skutecznie wykonywać takie zadania. Osobiście uważam to za sprzeczne z intuicją w tym przypadku.
Zauważ, że w (5) i (6), nadal ustawiamy kolumnę a, której nie wymagamy. Ale nie wiem, jak tego uniknąć. Jeśli filter() Funkcja miała argument, aby wybrać kolumny, które mają być zwrócone, możemy uniknąć tego problemu, ale wtedy funkcja nie wykona tylko jednego zadania (które jest również wyborem projektu dplyr).
Sub-assign by reference
Dplyr będzienigdy aktualizować przez odniesienie. Jest to kolejna ogromna (filozoficzna) różnica między tymi dwoma pakietami.
Na przykład, w data.tabela, którą możesz zrobić:
DT[a %in% some_vals, a := NA]
Która kolumna aktualizuje a przez odniesienie do tylko tych wierszy, które spełniają warunek. W tej chwili dplyr głęboko kopiuje całe dane.tabela wewnętrznie, aby dodać nową kolumnę. @BrodieG już o tym wspomniał w swojej odpowiedzi.
Ale głęboka Kopia może być zastąpiona płytką kopią, gdy FR #617 jest zaimplementowana. Również istotne: dplyr: FR#614. Zauważ, że modyfikowana kolumna zawsze będzie kopiowana (dlatego tad wolniejszy / mniej wydajny pamięci). Nie będzie możliwości aktualizacji kolumn według referencji.
Inne funkcje
W danych.tabela, można agregować podczas łączenia, a to jest bardziej proste do zrozumienia i jest wydajne w pamięci, ponieważ pośredni wynik połączenia nigdy nie jest zmaterializowany. Sprawdź ten post dla przykładu. Nie możesz (w tej chwili?) zrób to używając danych dplyr.tabela / dane.składnia ramki.
Data.tabela funkcja rolling joins nie jest również obsługiwana w składni dplyr.
-
Ostatnio zaimplementowaliśmy łączenie nakładek w danych.tabela do łączenia zakresów interwałów (oto przykład ), który jest obecnie oddzielną funkcją
foverlaps()i dlatego może być używany z operatorami rur (magrittr / pipeR? - nigdy nie próbowałem).Ale ostatecznie naszym celem jest zintegrowanie go z
[.data.table, abyśmy mogli zebrać inne funkcje, takie jak grupowanie, agregowanie podczas łączenia itp.. które będą miały te same ograniczenia opisane powyżej. -
Od 1.9.4, data.table implementuje automatyczne indeksowanie za pomocą klawiszy drugorzędnych do szybkiego wyszukiwania binarnego w oparciu o podzbiory na zwykłej składni R. Ex:
DT[x == 1]iDT[x %in% some_vals]automatycznie utworzą indeks przy pierwszym uruchomieniu, który następnie będzie używany na kolejnych podzbiorach z tej samej kolumny do szybkiego podzbioru za pomocą wyszukiwania binarnego. Ta funkcja będzie nadal ewoluować. Sprawdź ten gist dla krótki przegląd tej funkcji.Od sposobu implementacji {[5] } dla danych.tabel, nie korzysta z tej funkcji.
Cechą dplyr jest to, że zapewnia również interfejs do baz danych przy użyciu tej samej składni, które dane.stół w tej chwili nie ma.
Więc będziesz musiał rozważyć te (i prawdopodobnie inne punkty) i zdecydować na podstawie tego, czy te kompromisy są akceptowalne dla ty.
HTH
(1) zauważ, że wydajność pamięci bezpośrednio wpływa na szybkość (zwłaszcza gdy dane stają się większe), ponieważ wąskim gardłem w większości przypadków jest przenoszenie danych z pamięci głównej do pamięci podręcznej (i wykorzystanie danych w pamięci podręcznej w jak największym stopniu - zmniejszenie braków pamięci podręcznej - tak aby ograniczyć dostęp do pamięci głównej). Nie wdaję się w szczegóły.
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2017-05-23 12:18:25
Po prostu spróbuj.
library(rbenchmark)
library(dplyr)
library(data.table)
benchmark(
dplyr = diamondsDT %>%
filter(cut != "Fair") %>%
group_by(cut) %>%
summarize(AvgPrice = mean(price),
MedianPrice = as.numeric(median(price)),
Count = n()) %>%
arrange(desc(Count)),
data.table = diamondsDT[cut != "Fair",
list(AvgPrice = mean(price),
MedianPrice = as.numeric(median(price)),
Count = .N), by = cut][order(-Count)])[1:4]
Na ten problem wydaje się data.tabela jest 2,4 x szybsza niż dplyr przy użyciu danych.tabela:
test replications elapsed relative
2 data.table 100 2.39 1.000
1 dplyr 100 5.77 2.414
Poprawione na podstawie komentarza polimerazy.
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2014-12-16 22:13:24
Aby odpowiedzieć na twoje pytania:
- tak, używasz
data.table - ale nie tak efektywnie jak z czystą
data.tableskładnią
W wielu przypadkach będzie to akceptowalny kompromis dla tych, którzy chcą mieć składnię dplyr, choć prawdopodobnie będzie ona wolniejsza niż dplyr W przypadku zwykłych ramek danych.
Dużym czynnikiem wydaje się być to, że dplyr domyślnie kopiuje data.table podczas grupowania. Consider (using microbenchmark):
Unit: microseconds
expr min lq median
diamondsDT[, mean(price), by = cut] 3395.753 4039.5700 4543.594
diamondsDT[cut != "Fair"] 12315.943 15460.1055 16383.738
diamondsDT %>% group_by(cut) %>% summarize(AvgPrice = mean(price)) 9210.670 11486.7530 12994.073
diamondsDT %>% filter(cut != "Fair") 13003.878 15897.5310 17032.609
Filtrowanie jest o porównywalnej prędkości, ale zgrupowanie nie. uważam, że winowajcą jest ta linia w dplyr:::grouped_dt:
if (copy) {
data <- data.table::copy(data)
}
Gdzie copy domyślnie TRUE (i nie można łatwo zmienić na FALSE, które widzę). To prawdopodobnie nie stanowi 100% różnicy, ale ogólne koszty same w sobie na czymś wielkości diamonds najprawdopodobniej nie jest pełną różnicą.
Problem polega na tym, że aby mieć spójną gramatykę, dplyr wykonuje grupowanie w dwóch etapach. Najpierw ustawia klucze na kopii oryginalna Tabela danych, która pasuje do grup, a dopiero później grupuje. data.table po prostu przydziela pamięć dla największej grupy wyników, która w tym przypadku jest tylko jednym wierszem, więc to robi dużą różnicę w ilości pamięci, która musi być przydzielona.
Dla twojej wiadomości, Jeśli kogoś to obchodzi, znalazłem to używając treeprof (install_github("brodieg/treeprof")), jest to bardzo prosta i intuicyjna przeglądarka drzewa dla wyjścia Rprof:
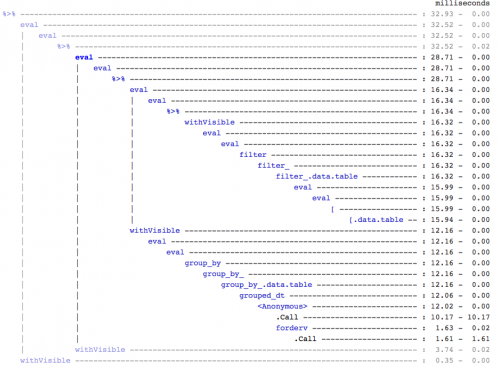
Uwaga powyższe jest obecnie działa tylko na macs AFAIK. Również, niestety, Rprof zapisuje wywołania typu packagename::funname jako anonimowe, więc może to być każde wywołanie {20]} wewnątrz {21]}, które są odpowiedzialne, ale z szybkich testów wyglądało to tak, jakby {22]} było tym dużym.
To powiedziawszy, możesz szybko zobaczyć, że nie ma zbyt wiele kosztów wokół wywołania [.data.table, ale istnieje również zupełnie oddzielna gałąź dla grupowania.
EDIT : aby potwierdzić kopiowanie:
> tracemem(diamondsDT)
[1] "<0x000000002747e348>"
> diamondsDT %>% group_by(cut) %>% summarize(AvgPrice = mean(price))
tracemem[0x000000002747e348 -> 0x000000002a624bc0]: <Anonymous> grouped_dt group_by_.data.table group_by_ group_by <Anonymous> freduce _fseq eval eval withVisible %>%
Source: local data table [5 x 2]
cut AvgPrice
1 Fair 4358.758
2 Good 3928.864
3 Very Good 3981.760
4 Premium 4584.258
5 Ideal 3457.542
> diamondsDT[, mean(price), by = cut]
cut V1
1: Ideal 3457.542
2: Premium 4584.258
3: Good 3928.864
4: Very Good 3981.760
5: Fair 4358.758
> untracemem(diamondsDT)
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2014-12-16 22:36:57