Jakie techniki istnieją w R do wizualizacji "macierzy odległości"?
Chciałbym przedstawić macierz odległości w artykule, który piszę i szukam dla niego dobrej wizualizacji.
Do tej pory natknąłem się na działki balonowe (użyłem go Tutaj, ale nie sądzę, żeby w tym przypadku zadziałało), heatmapy( tutaj ładny przykład , ale nie pozwalają na przedstawienie liczb w tabeli, popraw mnie, jeśli się mylę. Może połowa tabeli w kolorach i połowa z liczbami byłaby fajna) i na koniec elipsa korelacji wykresy (tu jest jakiś kod i przykład - fajnie jest użyć kształtu, ale nie jestem pewien jak go tutaj użyć).
Istnieją również różne metody klastrowania, ale będą one agregować dane (co jest , a nie, czego chcę), podczas gdy chcę przedstawić wszystkie dane.
Przykładowe dane:
nba <- read.csv("http://datasets.flowingdata.com/ppg2008.csv")
dist(nba[1:20, -1], )
7 answers
Tal, jest to szybki sposób na nałożenie tekstu na mapę cieplną. Należy zauważyć, że polega to na image, a nie heatmap, ponieważ ta ostatnia przesuwa Wykres, co utrudnia umieszczenie tekstu we właściwej pozycji.
Szczerze mówiąc, myślę, że ten wykres pokazuje zbyt dużo informacji, co sprawia, że jest trochę trudny do odczytania... możesz chcieć zapisać tylko określone wartości.
Inną szybszą opcją jest zapisanie wykresu jako pdf, zaimportowanie go do Inkscape (lub podobnego oprogramowania) i ręczne dodanie tekst w razie potrzeby.
Hope this helps
nba <- read.csv("http://datasets.flowingdata.com/ppg2008.csv")
dst <- dist(nba[1:20, -1],)
dst <- data.matrix(dst)
dim <- ncol(dst)
image(1:dim, 1:dim, dst, axes = FALSE, xlab="", ylab="")
axis(1, 1:dim, nba[1:20,1], cex.axis = 0.5, las=3)
axis(2, 1:dim, nba[1:20,1], cex.axis = 0.5, las=1)
text(expand.grid(1:dim, 1:dim), sprintf("%0.1f", dst), cex=0.6)
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2015-08-25 09:56:37
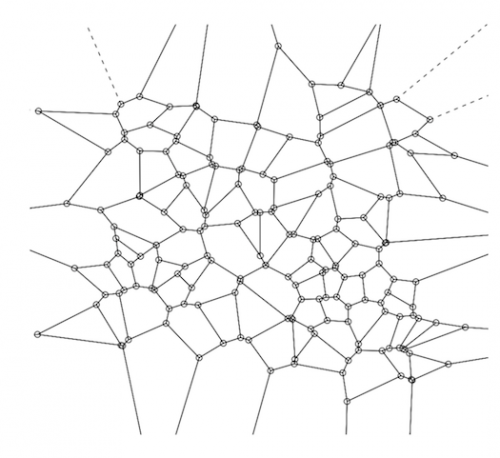
A Diagram Woronoi 'ego (Wykres rozkładu Woronoi' ego) jest jednym ze sposobów wizualnego przedstawienia macierzy odległości (DM).
Są również proste w tworzeniu i wykresach za pomocą R--możesz zrobić oba w jednej linii kodu R.
Jeśli nie jesteś zaznajomiony z tym aspektem geometrii obliczeniowej, relacja między nimi (VD & DM) jest prosta, chociaż krótkie podsumowanie może być pomocne.
Macierze odległości--tzn. macierz 2D pokazująca odległość między K-nearest neighbor, algorytm uczenia maszynowego, który przewiduje wartość danego punktu danych na podstawie średniej ważonej jego " K "najbliższych sąsiadów, w odległości mądry, gdzie" k " jest jakaś liczba całkowita, zwykle między 3 i 5.)
KNN jest koncepcyjnie bardzo prosty-każdy punkt danych w zestawie treningowym jest w istocie "pozycją" w jakiejś n-wymiarowej przestrzeni, więc następnym krokiem jest obliczenie odległości między każdym punktem A każdym innym punktem za pomocą pewnej metryki odległości (np. Euklidesa, Manhattana itp.). Podczas gdy etap treningowy-czyli konstruowanie macierzy odległości-jest prosty, używanie go do przewidywania wartości nowych punktów danych jest praktycznie obciążone przez pobieranie danych-znalezienie najbliższych 3 lub 4 punktów spośród kilku tysięcy lub kilku milionów rozproszonych w n-wymiarowej przestrzeni.
Dwie struktury danych są powszechnie używane do rozwiązania tego problemu: KD-trees i Voroni dekompozycje (tzw. "Tesselacja Dirichleta").
Rozkład Voronoi ' ego (VD) jest jednoznacznie określony przez macierz odległości-tzn. istnieje mapa 1: 1; więc rzeczywiście jest to wizualna reprezentacja macierzy odległości, chociaż ponownie, nie jest to ich cel-ich głównym celem jest efektywne przechowywanie danych używanych do przewidywania opartego na kNN.
Poza tym, czy jest to dobry pomysł, aby reprezentować matrycę odległości w ten sposób, prawdopodobnie zależy przede wszystkim od odbiorców. Dla większości, relacja między VD a macierzą odległości poprzedzającej nie będzie intuicyjna. Ale to nie znaczy, że jest niepoprawne-jeśli ktoś bez żadnego treningu statystycznego chciał wiedzieć, czy dwie populacje mają podobne rozkłady prawdopodobieństwa i pokazałeś im Wykres Q-Q, prawdopodobnie pomyślałby, że nie zająłeś się ich pytaniem. Tak więc dla tych, którzy wiedzą, na co patrzą, VD jest kompaktową, kompletną i dokładną reprezentacją DM.
Więc jak je zrobić?A Rozkład Voronoi konstruuje się poprzez wybranie (zwykle losowo) podzbioru punktów z zestawu treningowego (liczba ta różni się w zależności od okoliczności, ale jeśli mamy 1 000 000 punktów, to 100 jest rozsądną liczbą dla tego podzbioru). Te 100 punktów danych to Centra Voronoi ("VC").
Podstawową ideą rozkładu Voronoi jest to, że zamiast przesiać przez 1 000 000 punktów danych, aby znaleźć najbliższych sąsiadów, wystarczy spojrzeć na te 100, a gdy znajdziesz najbliższy VC, twoje poszukiwania najbliższych sąsiadów ograniczają się tylko do punktów w komórce Voronoi. Następnie dla każdego punktu danych w zestawie treningowym Oblicz VC, do którego jest najbliżej. Na koniec, dla każdego VC i powiązanych z nim punktów, Oblicz wypukły kadłub -- koncepcyjnie, tylko zewnętrzną granicę utworzoną przez przypisane punkty VC, które są najdalej od VC. Ten wypukły kadłub wokół centrum Woronoi tworzy " komórkę Woronoi."Kompletny VD jest wynikiem zastosowania tych trzy kroki do każdego VC w zestawie treningowym. Dzięki temu uzyskasz idealną tesselację powierzchni(patrz schemat poniżej).
Aby obliczyć VD w R, użyj pakietu tripack . Kluczową funkcją jest " voronoi.mozaika", do której po prostu przekazujesz we współrzędnych x i y osobno-surowe dane, a nie DM - wtedy możesz po prostu przekazać voronoi.mozaika do "działki".
library(tripack)
plot(voronoi.mosaic(runif(100), runif(100), duplicate="remove"))
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2015-08-25 09:52:03
Można również użyć algorytmów rysowania grafu skierowanego siłą do wizualizacji macierzy odległości, np.
nba <- read.csv("http://datasets.flowingdata.com/ppg2008.csv")
dist_m <- as.matrix(dist(nba[1:20, -1]))
dist_mi <- 1/dist_m # one over, as qgraph takes similarity matrices as input
library(qgraph)
jpeg('example_forcedraw.jpg', width=1000, height=1000, unit='px')
qgraph(dist_mi, layout='spring', vsize=3)
dev.off()
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2016-06-15 10:34:38
Możesz rozważyć rzut 2-d swojej macierzy (skalowanie wielowymiarowe). Oto link do tego jak to zrobić w R .
W przeciwnym razie, myślę, że jesteś na dobrej drodze z heatmap. Możesz dodawać swoje liczby bez większych trudności. Na przykład, budowanie off Learn R :library(ggplot2)
library(plyr)
library(arm)
library(reshape2)
nba <- read.csv("http://datasets.flowingdata.com/ppg2008.csv")
nba$Name <- with(nba, reorder(Name, PTS))
nba.m <- melt(nba)
nba.m <- ddply(nba.m, .(variable), transform,
rescale = rescale(value))
(p <- ggplot(nba.m, aes(variable, Name)) + geom_tile(aes(fill = rescale),
colour = "white") + scale_fill_gradient(low = "white",
high = "steelblue")+geom_text(aes(label=round(rescale,1))))
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2015-08-25 10:02:36
Przydatny może być dendrogram oparty na hierarchicznej analizie klastrów: http://www.statmethods.net/advstats/cluster.html
Wielowymiarowa analiza skalowania 2-D lub 3-D W R: http://www.statmethods.net/advstats/mds.html
Jeśli chcesz przejść do 3 + wymiarów, możesz chcieć poznać ggobi / rggobi: http://www.ggobi.org/rggobi/
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2010-06-21 03:32:59
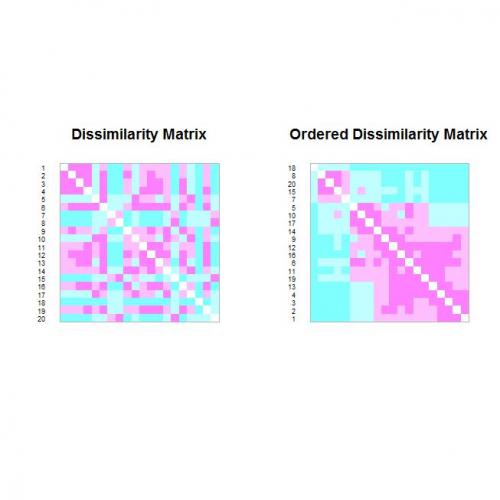
W książce" Numerical Ecology " Borcard et al. W 2011 roku zastosowano funkcję o nazwie *coldiss.r * znajdziesz go tutaj: http://ichthyology.usm.edu/courses/multivariate/coldiss.R
Koduje odległości, a nawet porządkuje rekordy przez odmienność.
Kolejnym dobrym pakietem będzie pakietseriation .
Odniesienie: Borcard, D., Gillet, F. & Legendre, P. (2011). Numerical Ecology with R. Springer.
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2014-01-03 04:29:41
Rozwiązanie wykorzystujące skalowanie wielowymiarowe
data = read.csv("http://datasets.flowingdata.com/ppg2008.csv", sep = ",")
dst = tcrossprod(as.matrix(data[,-1]))
dst = matrix(rep(diag(dst), 50L), ncol = 50L, byrow = TRUE) +
matrix(rep(diag(dst), 50L), ncol = 50L, byrow = FALSE) - 2*dst
library(MASS)
mds = isoMDS(dst)
#remove {type = "n"} to see dots
plot(mds$points, type = "n", pch = 20, cex = 3, col = adjustcolor("black", alpha = 0.3), xlab = "X", ylab = "Y")
text(mds$points, labels = rownames(data), cex = 0.75)
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2016-07-05 13:28:13



