Jak mogę poprawić wykrywanie łapek?
Po moim poprzednim pytaniu o znalezienie palców w każdej łapie, zacząłem ładować inne pomiary, aby zobaczyć, jak to wytrzyma. Niestety, szybko natknąłem się na problem z jednym z poprzednich kroków: rozpoznawanie łapek.
Widzisz, mój proof of concept w zasadzie wziął maksymalne ciśnienie każdego czujnika w czasie i zaczął szukać sumy każdego rzędu, dopóki nie znajdzie na tym != 0.0. Wtedy robi to samo dla kolumn i jak tylko znajdzie więcej niż 2 rzędy z tym znowu zero. Przechowuje minimalne i maksymalne wartości wierszy i kolumn do pewnego indeksu.
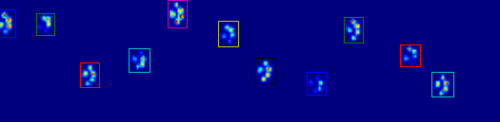
Jak widać na rysunku, działa to całkiem dobrze w większości przypadków. Jednak jest wiele wad tego podejścia (poza byciem bardzo prymitywnym): {]}
-
Ludzie mogą mieć "puste stopy", co oznacza, że istnieje kilka pustych rzędów w samym footprint. Ponieważ obawiałam się, że może się to zdarzyć również z (dużymi) psami, czekałam na co najmniej 2 lub 3 puste rzędy przed odcięciem łapy.
Tworzy to problem, jeśli inny kontakt wykonany w innej kolumnie, zanim dotrze do kilku pustych wierszy, rozszerzając tym samym obszar. Myślę, że mogę porównać kolumny i zobaczyć, czy przekraczają pewną wartość, muszą być oddzielne łapy.
-
Problem pogarsza się, gdy pies jest bardzo mały lub chodzi w wyższym tempie. To, co się dzieje, to to, że palce przedniej łapy wciąż się stykają, podczas gdy palce tylnej łapy zaczynają się aby nawiązać kontakt w tym samym obszarze co przednia łapa!
Z moim prostym skryptem nie będzie w stanie rozdzielić tych dwóch, ponieważ musiałby określić, które klatki tego obszaru należą do której łapy, podczas gdy obecnie musiałbym tylko patrzeć na maksymalne wartości wszystkich klatek.
Przykłady tego, gdzie zaczyna się źle:
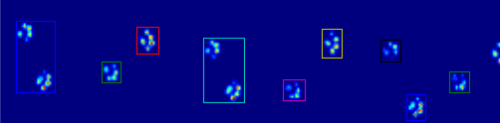

Więc teraz szukam lepszego sposobu rozpoznawania i oddzielania łap (po czym będę za problem decydowania, która to łapa!).
Update:
Majstrowałem, aby dostać Joe ' s (awesome!) odpowiedź zaimplementowana, ale mam trudności z wyodrębnieniem rzeczywistych danych paw z moich plików.

Coded_paws pokazuje mi wszystkie różne łapy, po nałożeniu na obraz maksymalnego nacisku(patrz wyżej). Jednak rozwiązanie przechodzi nad każdą ramką (aby oddzielić nakładające się łapy) i ustawia cztery atrybuty prostokąta, takie jak współrzędne lub wysokość / szerokość.
Nie mogę wymyślić, jak wziąć te atrybuty i zapisać je w jakiejś zmiennej, którą mogę zastosować do danych pomiarowych. Ponieważ muszę wiedzieć dla każdej łapy, jakie jest jej położenie podczas których klatek i połączyć to do której łapy jest (przód / tył, lewo / prawo).
Więc jak mogę użyć atrybutów prostokątów, aby wyodrębnić te wartości dla każdej łapy?
Mam pomiary, których użyłem w ustawieniu pytania w moim publicznym folderze Dropbox (przykład 1, przykład 2, przykład 3 ). dla wszystkich zainteresowanych założyłem również bloga , aby być na bieżąco: -)
3 answers
Jeśli chcesz tylko (częściowo) sąsiadujących ze sobą regionów, istnieje już łatwa implementacja w Pythonie: SciPy ' S ndimage.morfologia moduł. Jest to dość częsta operacja morfologii obrazu.
Zasadniczo masz 5 kroków:
def find_paws(data, smooth_radius=5, threshold=0.0001):
data = sp.ndimage.uniform_filter(data, smooth_radius)
thresh = data > threshold
filled = sp.ndimage.morphology.binary_fill_holes(thresh)
coded_paws, num_paws = sp.ndimage.label(filled)
data_slices = sp.ndimage.find_objects(coded_paws)
return object_slices
Rozmyj dane wejściowe, aby upewnić się, że łapy mają ciągły ślad. (Bardziej efektywne byłoby użycie większego jądra (
structurekwarg do różnych funkcjiscipy.ndimage.morphology) ale to nie działa właściwie z jakiegoś powodu...)Próg tablicy tak, że masz boolean tablicy miejsc, gdzie ciśnienie jest ponad jakąś wartość progową (tj.
thresh = data > value)Wypełnij wszelkie wewnętrzne otwory, aby mieć czystsze regiony (
filled = sp.ndimage.morphology.binary_fill_holes(thresh))Znajdź oddzielne sąsiadujące regiony (
coded_paws, num_paws = sp.ndimage.label(filled)). Zwraca tablicę z regionami zakodowanymi przez numer (Każdy region jest przylegającym obszarem unikalnej liczby całkowitej (1 do liczby łapki) z zerami wszędzie indziej)).Wyizoluj sąsiadujące ze sobą regiony za pomocą
data_slices = sp.ndimage.find_objects(coded_paws). Zwraca listę krotek obiektówslice, dzięki czemu można uzyskać region danych dla każdej paw z[data[x] for x in data_slices]. Zamiast tego narysujemy prostokąt na podstawie tych plastrów, co wymaga nieco więcej pracy.
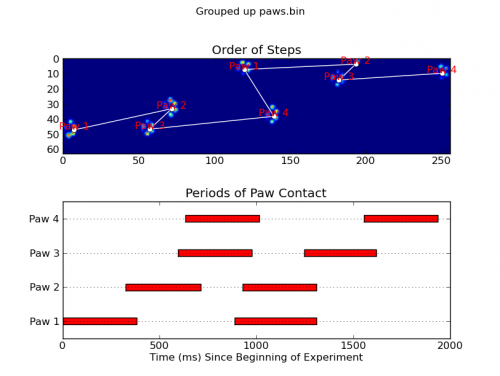
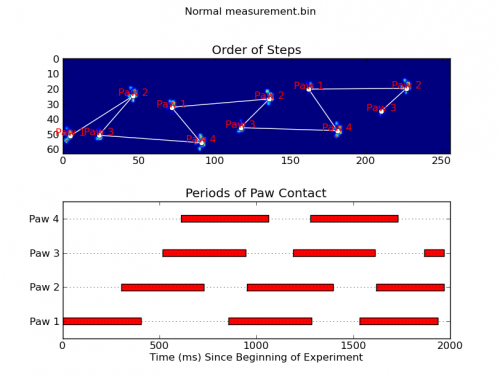
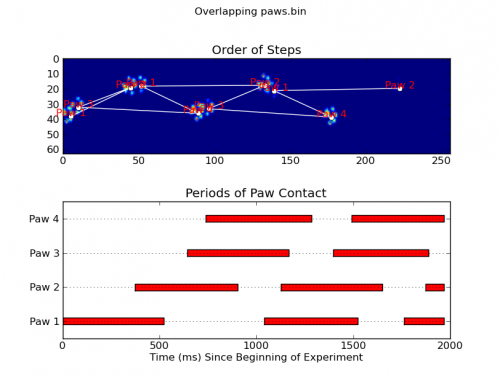
Dwie animacje poniżej pokazują przykładowe dane" nakładających się łap "i" pogrupowanych łap". Ta metoda wydaje się działać idealnie. (I o ile to coś warte, działa to znacznie płynniej niż poniższe obrazy GIF na moim komputerze, więc algorytm wykrywania łapek jest dość szybki...)
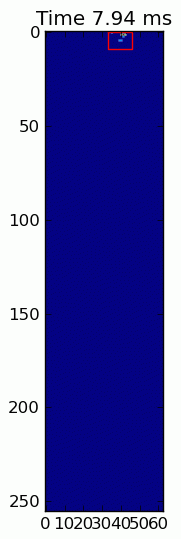
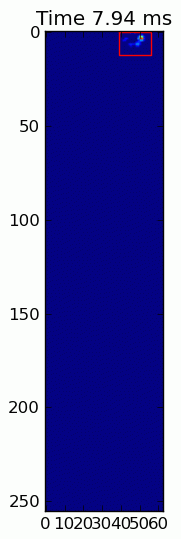
Oto pełny przykład (teraz ze znacznie bardziej szczegółowymi wyjaśnieniami). Zdecydowana większość z nich to odczytywanie danych wejściowych i tworzenie animacji. Rzeczywiste wykrywanie łapek to tylko 5 linijek kodu.
import numpy as np
import scipy as sp
import scipy.ndimage
import matplotlib.pyplot as plt
from matplotlib.patches import Rectangle
def animate(input_filename):
"""Detects paws and animates the position and raw data of each frame
in the input file"""
# With matplotlib, it's much, much faster to just update the properties
# of a display object than it is to create a new one, so we'll just update
# the data and position of the same objects throughout this animation...
infile = paw_file(input_filename)
# Since we're making an animation with matplotlib, we need
# ion() instead of show()...
plt.ion()
fig = plt.figure()
ax = fig.add_subplot(111)
fig.suptitle(input_filename)
# Make an image based on the first frame that we'll update later
# (The first frame is never actually displayed)
im = ax.imshow(infile.next()[1])
# Make 4 rectangles that we can later move to the position of each paw
rects = [Rectangle((0,0), 1,1, fc='none', ec='red') for i in range(4)]
[ax.add_patch(rect) for rect in rects]
title = ax.set_title('Time 0.0 ms')
# Process and display each frame
for time, frame in infile:
paw_slices = find_paws(frame)
# Hide any rectangles that might be visible
[rect.set_visible(False) for rect in rects]
# Set the position and size of a rectangle for each paw and display it
for slice, rect in zip(paw_slices, rects):
dy, dx = slice
rect.set_xy((dx.start, dy.start))
rect.set_width(dx.stop - dx.start + 1)
rect.set_height(dy.stop - dy.start + 1)
rect.set_visible(True)
# Update the image data and title of the plot
title.set_text('Time %0.2f ms' % time)
im.set_data(frame)
im.set_clim([frame.min(), frame.max()])
fig.canvas.draw()
def find_paws(data, smooth_radius=5, threshold=0.0001):
"""Detects and isolates contiguous regions in the input array"""
# Blur the input data a bit so the paws have a continous footprint
data = sp.ndimage.uniform_filter(data, smooth_radius)
# Threshold the blurred data (this needs to be a bit > 0 due to the blur)
thresh = data > threshold
# Fill any interior holes in the paws to get cleaner regions...
filled = sp.ndimage.morphology.binary_fill_holes(thresh)
# Label each contiguous paw
coded_paws, num_paws = sp.ndimage.label(filled)
# Isolate the extent of each paw
data_slices = sp.ndimage.find_objects(coded_paws)
return data_slices
def paw_file(filename):
"""Returns a iterator that yields the time and data in each frame
The infile is an ascii file of timesteps formatted similar to this:
Frame 0 (0.00 ms)
0.0 0.0 0.0
0.0 0.0 0.0
Frame 1 (0.53 ms)
0.0 0.0 0.0
0.0 0.0 0.0
...
"""
with open(filename) as infile:
while True:
try:
time, data = read_frame(infile)
yield time, data
except StopIteration:
break
def read_frame(infile):
"""Reads a frame from the infile."""
frame_header = infile.next().strip().split()
time = float(frame_header[-2][1:])
data = []
while True:
line = infile.next().strip().split()
if line == []:
break
data.append(line)
return time, np.array(data, dtype=np.float)
if __name__ == '__main__':
animate('Overlapping paws.bin')
animate('Grouped up paws.bin')
animate('Normal measurement.bin')
Aktualizacja: jeśli chodzi o określenie, która łapa jest w kontakcie z czujnikiem w jakich godzinach, najprostszym rozwiązaniem jest po prostu zrobić tę samą analizę, ale wykorzystać wszystkie dane na raz. (tzn. umieścić wejście w tablicy 3D i pracować z nią, zamiast poszczególnych przedziałów czasowych.) Ponieważ funkcje Ndimage SciPy ' ego są przeznaczone do pracy z tablicami n-wymiarowymi, nie musimy w ogóle modyfikować oryginalnej funkcji znajdującej łapę.
# This uses functions (and imports) in the previous code example!!
def paw_regions(infile):
# Read in and stack all data together into a 3D array
data, time = [], []
for t, frame in paw_file(infile):
time.append(t)
data.append(frame)
data = np.dstack(data)
time = np.asarray(time)
# Find and label the paw impacts
data_slices, coded_paws = find_paws(data, smooth_radius=4)
# Sort by time of initial paw impact... This way we can determine which
# paws are which relative to the first paw with a simple modulo 4.
# (Assuming a 4-legged dog, where all 4 paws contacted the sensor)
data_slices.sort(key=lambda dat_slice: dat_slice[2].start)
# Plot up a simple analysis
fig = plt.figure()
ax1 = fig.add_subplot(2,1,1)
annotate_paw_prints(time, data, data_slices, ax=ax1)
ax2 = fig.add_subplot(2,1,2)
plot_paw_impacts(time, data_slices, ax=ax2)
fig.suptitle(infile)
def plot_paw_impacts(time, data_slices, ax=None):
if ax is None:
ax = plt.gca()
# Group impacts by paw...
for i, dat_slice in enumerate(data_slices):
dx, dy, dt = dat_slice
paw = i%4 + 1
# Draw a bar over the time interval where each paw is in contact
ax.barh(bottom=paw, width=time[dt].ptp(), height=0.2,
left=time[dt].min(), align='center', color='red')
ax.set_yticks(range(1, 5))
ax.set_yticklabels(['Paw 1', 'Paw 2', 'Paw 3', 'Paw 4'])
ax.set_xlabel('Time (ms) Since Beginning of Experiment')
ax.yaxis.grid(True)
ax.set_title('Periods of Paw Contact')
def annotate_paw_prints(time, data, data_slices, ax=None):
if ax is None:
ax = plt.gca()
# Display all paw impacts (sum over time)
ax.imshow(data.sum(axis=2).T)
# Annotate each impact with which paw it is
# (Relative to the first paw to hit the sensor)
x, y = [], []
for i, region in enumerate(data_slices):
dx, dy, dz = region
# Get x,y center of slice...
x0 = 0.5 * (dx.start + dx.stop)
y0 = 0.5 * (dy.start + dy.stop)
x.append(x0); y.append(y0)
# Annotate the paw impacts
ax.annotate('Paw %i' % (i%4 +1), (x0, y0),
color='red', ha='center', va='bottom')
# Plot line connecting paw impacts
ax.plot(x,y, '-wo')
ax.axis('image')
ax.set_title('Order of Steps')
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2010-11-20 15:50:28
Nie jestem ekspertem w wykrywaniu obrazów i nie znam Pythona, ale dam sobie z tym radę...
Aby wykryć pojedyncze łapy, należy najpierw wybrać wszystko, co ma ciśnienie większe niż mały próg, bardzo bliskie żadnemu ciśnieniu. Każdy piksel / punkt, który znajduje się powyżej tego powinien być " zaznaczony."Następnie każdy piksel sąsiadujący ze wszystkimi "zaznaczonymi" pikselami zostaje oznaczony, a proces ten powtarza się kilka razy. Masy, które są całkowicie połączone, będą formowane, więc masz odrębny obiektów. Następnie każdy "obiekt" ma minimalną i maksymalną wartość x i y, więc obwiedniowe pola mogą być starannie pakowane wokół nich.
Pseudokod:
(MARK) ALL PIXELS ABOVE (0.5)
(MARK) ALL PIXELS (ADJACENT) TO (MARK) PIXELS
REPEAT (STEP 2) (5) TIMES
SEPARATE EACH TOTALLY CONNECTED MASS INTO A SINGLE OBJECT
MARK THE EDGES OF EACH OBJECT, AND CUT APART TO FORM SLICES.
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2010-11-03 21:42:29
Uwaga: mówię piksel, ale to mogą być regiony używające średniej pikseli. Kolejną kwestią jest optymalizacja...
Wygląda na to, że trzeba przeanalizować funkcję (ciśnienie w czasie) dla każdego piksela i określić gdzie funkcja obraca się (gdy zmienia się > X w przeciwnym kierunku, jest to uważane za zwrot do błędów licznika).
Jeśli wiesz, w jakich klatkach się obraca, będziesz wiedział, w której ramie ciśnienie było największe i będziesz wiedział, gdzie było to najmniej twardy między dwoma łapami. Teoretycznie poznałbyś wtedy dwie klatki, w których łapy naciskały najbardziej mocno i możesz obliczyć średnią z tych interwałów.
Po czym przejdę do problemu decydowania, która to łapa!
Jest to ta sama trasa, co wcześniej, wiedząc, kiedy każda łapa przykłada największą presję pomaga podjąć decyzję.
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2010-11-04 01:09:24