Usuń wiersze z wszystkimi lub niektórymi serwerami NAs (brakującymi wartościami) w danych.ramka
Chciałbym usunąć linie w tej ramce danych, które:
A) zawiera NAs we wszystkich kolumnach. poniżej znajduje się moja przykładowa ramka danych.
gene hsap mmul mmus rnor cfam
1 ENSG00000208234 0 NA NA NA NA
2 ENSG00000199674 0 2 2 2 2
3 ENSG00000221622 0 NA NA NA NA
4 ENSG00000207604 0 NA NA 1 2
5 ENSG00000207431 0 NA NA NA NA
6 ENSG00000221312 0 1 2 3 2
Zasadniczo chciałbym uzyskać ramkę danych, taką jak poniżej.
gene hsap mmul mmus rnor cfam
2 ENSG00000199674 0 2 2 2 2
6 ENSG00000221312 0 1 2 3 2
B) zawiera NA s tylko w niektórych kolumnach , więc mogę również uzyskać ten wynik:
gene hsap mmul mmus rnor cfam
2 ENSG00000199674 0 2 2 2 2
4 ENSG00000207604 0 NA NA 1 2
6 ENSG00000221312 0 1 2 3 2
15 answers
Zobacz też complete.cases :
> final[complete.cases(final), ]
gene hsap mmul mmus rnor cfam
2 ENSG00000199674 0 2 2 2 2
6 ENSG00000221312 0 1 2 3 2
na.omit pozwala na częściowy wybór poprzez włączenie tylko niektórych kolumn ramki danych:
> final[complete.cases(final[ , 5:6]),]
gene hsap mmul mmus rnor cfam
2 ENSG00000199674 0 2 2 2 2
4 ENSG00000207604 0 NA NA 1 2
6 ENSG00000221312 0 1 2 3 2
is.na, musisz zrobić coś w stylu:
> final[rowSums(is.na(final[ , 5:6])) == 0, ]
gene hsap mmul mmus rnor cfam
2 ENSG00000199674 0 2 2 2 2
4 ENSG00000207604 0 NA NA 1 2
6 ENSG00000221312 0 1 2 3 2
Ale używanie complete.cases jest dużo bardziej przejrzyste i szybsze.
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2017-06-14 15:10:51
Spróbuj na.omit(your.data.frame). Jeśli chodzi o drugie pytanie, spróbuj opublikować je jako inne pytanie(dla jasności).
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2011-02-01 12:00:44
Preferuję następujący sposób sprawdzania, czy wiersze zawierają dowolny NAs:
row.has.na <- apply(final, 1, function(x){any(is.na(x))})
Zwraca wektor logiczny z wartościami określającymi, czy w wierszu jest jakieś NA. Możesz go użyć, aby zobaczyć, ile wierszy będziesz musiał upuścić:
sum(row.has.na)
I w końcu je porzucić
final.filtered <- final[!row.has.na,]
W przypadku filtrowania wierszy z określoną częścią serwera NAs staje się to nieco trudniejsze (na przykład możesz podać 'final [, 5:6]' do 'apply'). Generalnie rozwiązanie Jorisa Meysa wydaje się bardziej eleganckie.
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2011-02-02 21:58:33
Jeśli lubisz rury (%>%), tidyr's Nowy drop_na jest twoim przyjacielem:
library(tidyr)
df %>% drop_na()
# gene hsap mmul mmus rnor cfam
# 2 ENSG00000199674 0 2 2 2 2
# 6 ENSG00000221312 0 1 2 3 2
df %>% drop_na(rnor, cfam)
# gene hsap mmul mmus rnor cfam
# 2 ENSG00000199674 0 2 2 2 2
# 4 ENSG00000207604 0 NA NA 1 2
# 6 ENSG00000221312 0 1 2 3 2
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2016-08-16 08:49:23
Inną opcją, jeśli chcesz mieć większą kontrolę nad tym, jak wiersze są uważane za nieprawidłowe, jest
final <- final[!(is.na(final$rnor)) | !(is.na(rawdata$cfam)),]
Używając powyższego, to:
gene hsap mmul mmus rnor cfam
1 ENSG00000208234 0 NA NA NA 2
2 ENSG00000199674 0 2 2 2 2
3 ENSG00000221622 0 NA NA 2 NA
4 ENSG00000207604 0 NA NA 1 2
5 ENSG00000207431 0 NA NA NA NA
6 ENSG00000221312 0 1 2 3 2
Staje się:
gene hsap mmul mmus rnor cfam
1 ENSG00000208234 0 NA NA NA 2
2 ENSG00000199674 0 2 2 2 2
3 ENSG00000221622 0 NA NA 2 NA
4 ENSG00000207604 0 NA NA 1 2
6 ENSG00000221312 0 1 2 3 2
...gdzie usuwa się tylko wiersz 5, ponieważ jest to jedyny wiersz zawierający NAs zarówno dla rnor, jak i cfam. Logika logiczna może być następnie zmieniona w celu dopasowania do konkretnych wymagań.
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2013-11-05 06:30:24
Jeśli chcesz kontrolować, ile serwerów NAs jest ważnych dla każdego wiersza, wypróbuj tę funkcję. W przypadku wielu zestawów danych ankietowych zbyt wiele pustych odpowiedzi na pytania może zrujnować wyniki. Są więc usuwane po pewnym progu. Ta funkcja pozwoli Ci wybrać, ile NAs może mieć wiersz przed jego usunięciem:
delete.na <- function(DF, n=0) {
DF[rowSums(is.na(DF)) <= n,]
}
Domyślnie eliminuje wszystkie NAs:
delete.na(final)
gene hsap mmul mmus rnor cfam
2 ENSG00000199674 0 2 2 2 2
6 ENSG00000221312 0 1 2 3 2
Lub określ maksymalną dozwoloną liczbę NAs:
delete.na(final, 2)
gene hsap mmul mmus rnor cfam
2 ENSG00000199674 0 2 2 2 2
4 ENSG00000207604 0 NA NA 1 2
6 ENSG00000221312 0 1 2 3 2
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2016-11-18 13:59:11
Zwróci wiersze, które mają co najmniej jedną wartość inną niż NA.
final[rowSums(is.na(final))<length(final),]
Zwróci wiersze, które mają co najmniej dwie wartości inne niż NA.
final[rowSums(is.na(final))<(length(final)-1),]
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2014-09-19 14:39:08
Używając pakietu dplyr możemy filtrować NA w następujący sposób:
dplyr::filter(df, !is.na(columnname))
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2017-04-12 05:44:40
Możemy również użyć funkcji podzbioru do tego celu.
finalData<-subset(data,!(is.na(data["mmul"]) | is.na(data["rnor"])))
To da tylko te wiersze, które nie mają NA zarówno w mmul jak i rnor
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2014-11-11 22:20:38
Jeśli wydajność jest priorytetem, użyj data.table i na.omit() z opcjonalnym param cols=.
na.omit.data.table jest najszybszy na moim benchmarku (patrz poniżej), czy dla wszystkich kolumn, czy dla wybranych kolumn (pytanie OP część 2).
Jeśli nie chcesz używać data.table, Użyj complete.cases().
Na wanilii data.frame, complete.cases jest szybszy niż na.omit() lub dplyr::drop_na(). Zauważ, że na.omit.data.frame nie obsługuje cols=.
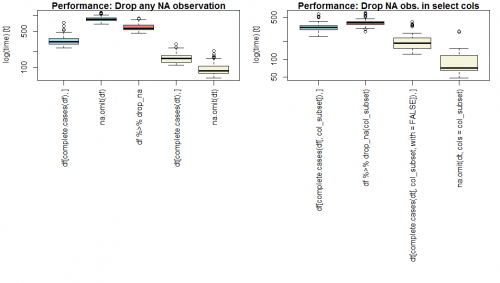
Benchmark result
Oto porównanie base (blue), dplyr (pink), and data.table (yellow) methods for dropping or select missing observations, on mentional dataset of 1 million observations of 20 numeric variables with independent 5% likely of being missing, and a podzbiór of 4 variables for part 2.
Wyniki mogą się różnić w zależności od długości, szerokości i sparsity konkretnego zbioru danych.
Uwaga skala dziennika na osi y.
Benchmark script
#------- Adjust these assumptions for your own use case ------------
row_size <- 1e6L
col_size <- 20 # not including ID column
p_missing <- 0.05 # likelihood of missing observation (except ID col)
col_subset <- 18:21 # second part of question: filter on select columns
#------- System info for benchmark ----------------------------------
R.version # R version 3.4.3 (2017-11-30), platform = x86_64-w64-mingw32
library(data.table); packageVersion('data.table') # 1.10.4.3
library(dplyr); packageVersion('dplyr') # 0.7.4
library(tidyr); packageVersion('tidyr') # 0.8.0
library(microbenchmark)
#------- Example dataset using above assumptions --------------------
fakeData <- function(m, n, p){
set.seed(123)
m <- matrix(runif(m*n), nrow=m, ncol=n)
m[m<p] <- NA
return(m)
}
df <- cbind( data.frame(id = paste0('ID',seq(row_size)),
stringsAsFactors = FALSE),
data.frame(fakeData(row_size, col_size, p_missing) )
)
dt <- data.table(df)
par(las=3, mfcol=c(1,2), mar=c(22,4,1,1)+0.1)
boxplot(
microbenchmark(
df[complete.cases(df), ],
na.omit(df),
df %>% drop_na,
dt[complete.cases(dt), ],
na.omit(dt)
), xlab='',
main = 'Performance: Drop any NA observation',
col=c(rep('lightblue',2),'salmon',rep('beige',2))
)
boxplot(
microbenchmark(
df[complete.cases(df[,col_subset]), ],
#na.omit(df), # col subset not supported in na.omit.data.frame
df %>% drop_na(col_subset),
dt[complete.cases(dt[,col_subset,with=FALSE]), ],
na.omit(dt, cols=col_subset) # see ?na.omit.data.table
), xlab='',
main = 'Performance: Drop NA obs. in select cols',
col=c('lightblue','salmon',rep('beige',2))
)
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2018-02-16 15:47:40
Na twoje pierwsze pytanie, Mam kod, który mi odpowiada, aby pozbyć się wszystkich NAs. Dzięki za @Gregor żeby było prościej.
final[!(rowSums(is.na(final))),]
Dla drugiego pytania, kod jest tylko alternatywą od poprzedniego rozwiązania.
final[as.logical((rowSums(is.na(final))-5)),]
Zauważ, że -5 to liczba kolumn w Twoich danych. Spowoduje to wyeliminowanie wierszy ze wszystkimi NAs, ponieważ sumy wierszy sumują się do 5 i stają się zerami po odjęciu. Tym razem as.logiczne jest konieczne.
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2018-03-07 14:57:40
Jestem syntezatorem:). Tutaj połączyłem odpowiedzi w jedną funkcję:
#' keep rows that have a certain number (range) of NAs anywhere/somewhere and delete others
#' @param df a data frame
#' @param col restrict to the columns where you would like to search for NA; eg, 3, c(3), 2:5, "place", c("place","age")
#' \cr default is NULL, search for all columns
#' @param n integer or vector, 0, c(3,5), number/range of NAs allowed.
#' \cr If a number, the exact number of NAs kept
#' \cr Range includes both ends 3<=n<=5
#' \cr Range could be -Inf, Inf
#' @return returns a new df with rows that have NA(s) removed
#' @export
ez.na.keep = function(df, col=NULL, n=0){
if (!is.null(col)) {
# R converts a single row/col to a vector if the parameter col has only one col
# see https://radfordneal.wordpress.com/2008/08/20/design-flaws-in-r-2-%E2%80%94-dropped-dimensions/#comments
df.temp = df[,col,drop=FALSE]
} else {
df.temp = df
}
if (length(n)==1){
if (n==0) {
# simply call complete.cases which might be faster
result = df[complete.cases(df.temp),]
} else {
# credit: http://stackoverflow.com/a/30461945/2292993
log <- apply(df.temp, 2, is.na)
logindex <- apply(log, 1, function(x) sum(x) == n)
result = df[logindex, ]
}
}
if (length(n)==2){
min = n[1]; max = n[2]
log <- apply(df.temp, 2, is.na)
logindex <- apply(log, 1, function(x) {sum(x) >= min && sum(x) <= max})
result = df[logindex, ]
}
return(result)
}
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2016-12-10 18:26:40
Zakładając dat jako ramkę danych, oczekiwany wynik można uzyskać za pomocą
1.rowSums
> dat[!rowSums((is.na(dat))),]
gene hsap mmul mmus rnor cfam
2 ENSG00000199674 0 2 2 2 2
6 ENSG00000221312 0 1 2 3 2
2.lapply
> dat[!Reduce('|',lapply(dat,is.na)),]
gene hsap mmul mmus rnor cfam
2 ENSG00000199674 0 2 2 2 2
6 ENSG00000221312 0 1 2 3 2
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2017-03-15 16:51:32
delete.dirt <- function(DF, dart=c('NA')) {
dirty_rows <- apply(DF, 1, function(r) !any(r %in% dart))
DF <- DF[dirty_rows, ]
}
mydata <- delete.dirt(mydata)
Powyższa funkcja usuwa wszystkie wiersze z ramki danych, która ma 'NA' w dowolnej kolumnie i zwraca wynikowe dane. Jeśli chcesz sprawdzić wiele wartości, takich jak NA i ? Zmień dart=c('NA') w funkcji param na dart=c('NA', '?')
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2018-02-22 22:19:46
Myślę, że to może być bardziej elegancko rozwiązane w ten sposób
m <- matrix(1:25, ncol = 5)
m[c(1, 6, 13, 25)] <- NA
df <- data.frame(m)
library(dplyr)
df %>%
filter_all(any_vars(is.na(.)))
#> X1 X2 X3 X4 X5
#> 1 NA NA 11 16 21
#> 2 3 8 NA 18 23
#> 3 5 10 15 20 NA
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2018-05-08 20:35:47