Wypełnianie luk w tablicy numpy
Chcę tylko interpolować, najprościej mówiąc, zestaw danych 3D. Interpolacja liniowa, najbliższy sąsiad, wszystko, co by wystarczyło (to jest uruchomienie jakiegoś algorytmu, więc nie jest wymagane dokładne oszacowanie).
W nowych wersjach scipy przydałyby się takie rzeczy jak griddata, ale obecnie mam tylko scipy 0.8. Mam więc tablicę" cube " (data[:,:,:], (NixNjxNk)) i tablicę Flag (flags[:,:,:,], True lub False) tego samego rozmiaru. Chcę interpolować moje dane dla elementy danych, w których odpowiadający im element znacznika jest False, używając np. najbliższego ważnego punktu datapoint w danych lub jakiejś liniowej kombinacji punktów "bliskich".
W zbiorze danych mogą występować duże luki w co najmniej dwóch wymiarach. Poza kodowaniem pełnowymiarowego algorytmu najbliższego sąsiada przy użyciu kdtrees lub podobnego, nie mogę znaleźć ogólnego, N-wymiarowego interpolatora najbliższego sąsiada.
5 answers
Możesz skonfigurować algorytm wzrostu kryształów zmieniający widok na przemian wzdłuż każdej osi, zastępując tylko dane, które są oznaczone False, ale mają True sąsiada. Daje to wynik podobny do "najbliższego sąsiada" (ale nie w odległości euklidesowej lub Manhattanu-myślę, że może to być najbliższy sąsiad, jeśli liczysz piksele, licząc wszystkie łączące się piksele ze wspólnymi narożnikami) powinno to być dość wydajne z NumPy, ponieważ iterauje tylko iteracje osi i zbieżności, a nie małe plastry danych.
import numpy as np
# -- setup --
shape = (10,10,10)
dim = len(shape)
data = np.random.random(shape)
flag = np.zeros(shape, dtype=bool)
t_ct = int(data.size/5)
flag.flat[np.random.randint(0, flag.size, t_ct)] = True
# True flags the data
# -- end setup --
slcs = [slice(None)]*dim
while np.any(~flag): # as long as there are any False's in flag
for i in range(dim): # do each axis
# make slices to shift view one element along the axis
slcs1 = slcs[:]
slcs2 = slcs[:]
slcs1[i] = slice(0, -1)
slcs2[i] = slice(1, None)
# replace from the right
repmask = np.logical_and(~flag[slcs1], flag[slcs2])
data[slcs1][repmask] = data[slcs2][repmask]
flag[slcs1][repmask] = True
# replace from the left
repmask = np.logical_and(~flag[slcs2], flag[slcs1])
data[slcs2][repmask] = data[slcs1][repmask]
flag[slcs2][repmask] = True
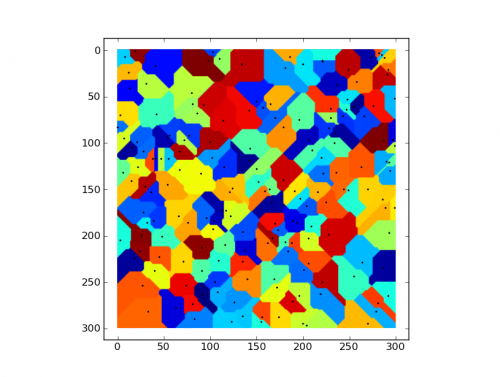
Na dobrą sprawę, oto wizualizacja (2D) stref obsadzonych przez dane pierwotnie oznaczone True.
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2011-04-05 18:58:10
Używając scipy.ndimage, twój problem można rozwiązać za pomocą interpolacji najbliższego sąsiada w 2 liniach:
from scipy import ndimage as nd
indices = nd.distance_transform_edt(invalid_cell_mask, return_distances=False, return_indices=True)
data = data[tuple(ind)]
Teraz w postaci funkcji:
import numpy as np
from scipy import ndimage as nd
def fill(data, invalid=None):
"""
Replace the value of invalid 'data' cells (indicated by 'invalid')
by the value of the nearest valid data cell
Input:
data: numpy array of any dimension
invalid: a binary array of same shape as 'data'.
data value are replaced where invalid is True
If None (default), use: invalid = np.isnan(data)
Output:
Return a filled array.
"""
if invalid is None: invalid = np.isnan(data)
ind = nd.distance_transform_edt(invalid,
return_distances=False,
return_indices=True)
return data[tuple(ind)]
Exemple użycia:
def test_fill(s,d):
# s is size of one dimension, d is the number of dimension
data = np.arange(s**d).reshape((s,)*d)
seed = np.zeros(data.shape,dtype=bool)
seed.flat[np.random.randint(0,seed.size,int(data.size/20**d))] = True
return fill(data,-seed), seed
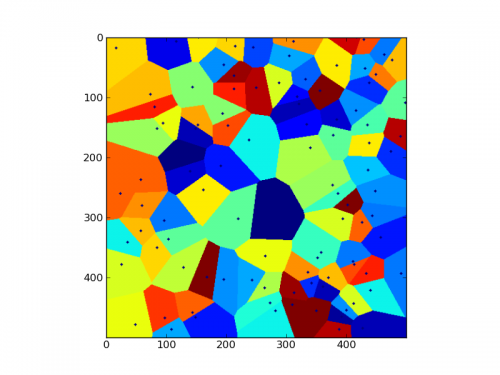
import matplotlib.pyplot as plt
data,seed = test_fill(500,2)
data[nd.binary_dilation(seed,iterations=2)] = 0 # draw (dilated) seeds in black
plt.imshow(np.mod(data,42)) # show cluster
Wynik:
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2015-02-03 07:42:06
Jakiś czas temu napisałem ten scenariusz do mojego doktoratu: https://github.com/Technariumas/Inpainting
Przykład: http://blog.technariumas.lt/post/117630308826/healing-holes-in-python-arrays
Powoli, ale robi robotę. Jądro Gaussa jest najlepszym wyborem, wystarczy sprawdzić rozmiar / wartości sigma.
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2015-04-30 21:59:10
Możesz spróbować rozwiązać swój problem jak:
# main ideas described in very high level pseudo code
choose suitable base kernel shape and type (gaussian?)
while true
loop over your array (moving average manner)
adapt your base kernel to current sparsity pattern
set current value based on adapted kernel
break if converged
W rzeczywistości można to zaimplementować w dość prosty sposób(zwłaszcza jeśli wydajność nie jest głównym problemem).
Oczywiście jest to tylko heurystyka i musisz zrobić kilka eksperymentów z rzeczywistymi danymi, aby znaleźć odpowiedni schemat adaptacyjny. Gdy widzisz adaptację jądra jako ponowne ważenie jądra, możesz chcieć to zrobić na podstawie tego, jak wartości zostały propagowane. Na przykład Twoje ciężary dla oryginalnych podpór to 1 i one rozpadów związanych z iteracją, na której się pojawiły.
Również określenie, kiedy proces ten rzeczywiście się zbiegł, może być trudne. W zależności od zastosowania może być rozsądne, aby pozostawić niektóre "regiony luki" pozostają "niewypełnione".Update : Oto bardzo prosta implementacja wzdłuż linii*) opisanych powyżej:
from numpy import any, asarray as asa, isnan, NaN, ones, seterr
from numpy.lib.stride_tricks import as_strided as ast
from scipy.stats import nanmean
def _a2t(a):
"""Array to tuple."""
return tuple(a.tolist())
def _view(D, shape, strides):
"""View of flattened neighbourhood of D."""
V= ast(D, shape= shape, strides= strides)
return V.reshape(V.shape[:len(D.shape)]+ (-1,))
def filler(A, n_shape, n_iter= 49):
"""Fill in NaNs from mean calculated from neighbour."""
# boundary conditions
D= NaN* ones(_a2t(asa(A.shape)+ asa(n_shape)- 1), dtype= A.dtype)
slc= tuple([slice(n/ 2, -(n/ 2)) for n in n_shape])
D[slc]= A
# neighbourhood
shape= _a2t(asa(D.shape)- asa(n_shape)+ 1)+ n_shape
strides= D.strides* 2
# iterate until no NaNs, but not more than n iterations
old= seterr(invalid= 'ignore')
for k in xrange(n_iter):
M= isnan(D[slc])
if not any(M): break
D[slc][M]= nanmean(_view(D, shape, strides), -1)[M]
seterr(**old)
A[:]= D[slc]
I prosta demonstracja filler(.) działania, byłaby czymś w rodzaju:
In []: x= ones((3, 6, 99))
In []: x.sum(-1)
Out[]:
array([[ 99., 99., 99., 99., 99., 99.],
[ 99., 99., 99., 99., 99., 99.],
[ 99., 99., 99., 99., 99., 99.]])
In []: x= NaN* x
In []: x[1, 2, 3]= 1
In []: x.sum(-1)
Out[]:
array([[ nan, nan, nan, nan, nan, nan],
[ nan, nan, nan, nan, nan, nan],
[ nan, nan, nan, nan, nan, nan]])
In []: filler(x, (3, 3, 5))
In []: x.sum(-1)
Out[]:
array([[ 99., 99., 99., 99., 99., 99.],
[ 99., 99., 99., 99., 99., 99.],
[ 99., 99., 99., 99., 99., 99.]])
*) więc tutaj {[4] } jest po prostu używane do zademonstrowania idei procesu adaptacji. Na podstawie tej demonstracji powinno być dość proste wdrożenie bardziej złożonego systemu ważenia adaptacyjnego i rozkładającego się. Należy również pamiętać, że nie zwraca się uwagi na rzeczywistą wydajność wykonania, ale nadal powinna być dobra (z rozsądnymi kształtami wejściowymi).
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2011-04-08 08:35:27
Może to, czego szukasz, to algorytm uczenia maszynowego, jak sieć neuronowa lub maszyna wektorowa.
Możesz sprawdzić tę stronę, która zawiera linki do pakietów SVM dla Pythona: http://web.media.mit.edu / ~stefie10/technical/pythonml.html
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2011-04-05 11:57:22