Jak mogę wykreślić 2 różne osi y?
Chciałbym nałożyć dwa wykresy rozrzutu w R tak, że każdy zestaw punktów ma swoją własną (inną) oś y (tj. w pozycjach 2 i 4 na rysunku), ale punkty pojawiają się nałożone na tej samej rysunku.
Czy można to zrobić z plot?
Edit przykładowy kod pokazujący problem
# example code for SO question
y1 <- rnorm(10, 100, 20)
y2 <- rnorm(10, 1, 1)
x <- 1:10
# in this plot y2 is plotted on what is clearly an inappropriate scale
plot(y1 ~ x, ylim = c(-1, 150))
points(y2 ~ x, pch = 2)
6 answers
Update: skopiowany materiał, który był na R wiki w http://rwiki.sciviews.org/doku.php?id=tips:graphics-base:2yaxes , link teraz uszkodzony: również dostępne z wayback machine
Dwie różne osie y na tej samej działce
(materiał autor: Daniel Rajdl 2006/03/31 15: 26)
Należy pamiętać, że jest bardzo niewiele sytuacji, w których właściwe jest użycie dwóch różnych skal na tej samej działce. Bardzo łatwo jest wprowadzić w błąd przeglądarka Grafiki. Sprawdź dwa poniższe przykłady i komentarze na ten temat (przykład1, example2 z Junk Charts ), a także ten artykuł Stephena Few (który stwierdza: "z pewnością nie mogę raz na zawsze stwierdzić, że wykresy z osiami o podwójnej skali nigdy nie są użyteczne; tyle, że nie mogę myśleć o sytuacji, która uzasadniałaby je w świetle innych, lepszych rozwiązań.") Zobacz też punkt #4 w tej kreskówce ...
Jeśli jeśli są określone, podstawową receptą jest utworzenie pierwszego wykresu, ustawienie par(new=TRUE), aby zapobiec wyczyszczeniu urządzenia graficznego przez R, utworzenie drugiego wykresu za pomocą axes=FALSE (i ustawienie xlab i ylab, aby były puste – ann=FALSE powinno również działać), a następnie użycie axis(side=4), aby dodać nową oś po prawej stronie, i mtext(...,side=4), aby dodać etykietę osi po prawej stronie. Oto przykład użycia odrobiny zmyślonych danych:
set.seed(101)
x <- 1:10
y <- rnorm(10)
## second data set on a very different scale
z <- runif(10, min=1000, max=10000)
par(mar = c(5, 4, 4, 4) + 0.3) # Leave space for z axis
plot(x, y) # first plot
par(new = TRUE)
plot(x, z, type = "l", axes = FALSE, bty = "n", xlab = "", ylab = "")
axis(side=4, at = pretty(range(z)))
mtext("z", side=4, line=3)
twoord.plot() w pakiecie plotrix automatyzuje ten proces, podobnie jak w pakiecie doubleYScale() ]} paczka.
Inny przykład (zaadaptowany z listy dyskusyjnej R przez Roberta W. Baera):
## set up some fake test data
time <- seq(0,72,12)
betagal.abs <- c(0.05,0.18,0.25,0.31,0.32,0.34,0.35)
cell.density <- c(0,1000,2000,3000,4000,5000,6000)
## add extra space to right margin of plot within frame
par(mar=c(5, 4, 4, 6) + 0.1)
## Plot first set of data and draw its axis
plot(time, betagal.abs, pch=16, axes=FALSE, ylim=c(0,1), xlab="", ylab="",
type="b",col="black", main="Mike's test data")
axis(2, ylim=c(0,1),col="black",las=1) ## las=1 makes horizontal labels
mtext("Beta Gal Absorbance",side=2,line=2.5)
box()
## Allow a second plot on the same graph
par(new=TRUE)
## Plot the second plot and put axis scale on right
plot(time, cell.density, pch=15, xlab="", ylab="", ylim=c(0,7000),
axes=FALSE, type="b", col="red")
## a little farther out (line=4) to make room for labels
mtext("Cell Density",side=4,col="red",line=4)
axis(4, ylim=c(0,7000), col="red",col.axis="red",las=1)
## Draw the time axis
axis(1,pretty(range(time),10))
mtext("Time (Hours)",side=1,col="black",line=2.5)
## Add Legend
legend("topleft",legend=c("Beta Gal","Cell Density"),
text.col=c("black","red"),pch=c(16,15),col=c("black","red"))
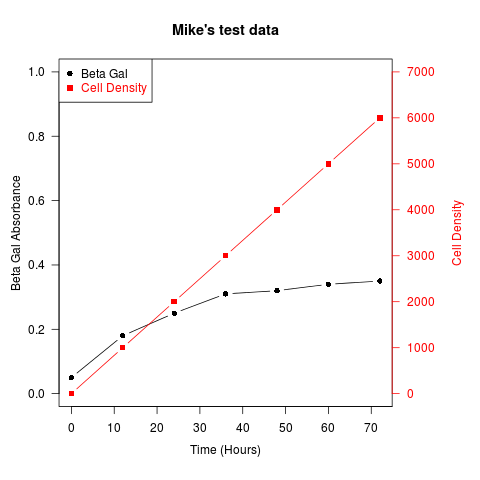
Podobne receptury mogą być stosowane do nakładania Wykresów różnych typów-wykresów słupkowych, histogramów itp..
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2014-12-16 23:28:05
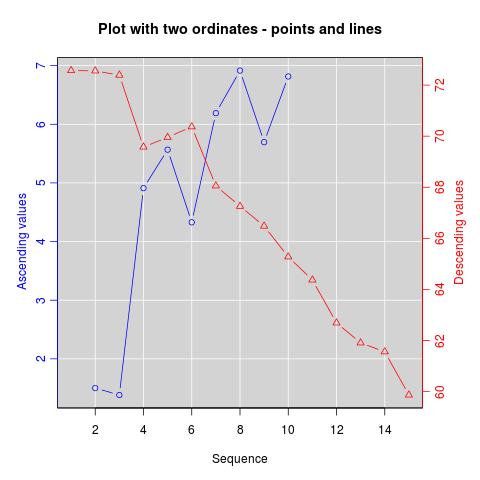
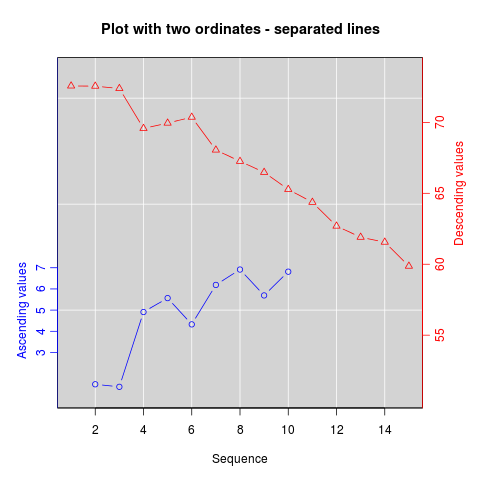
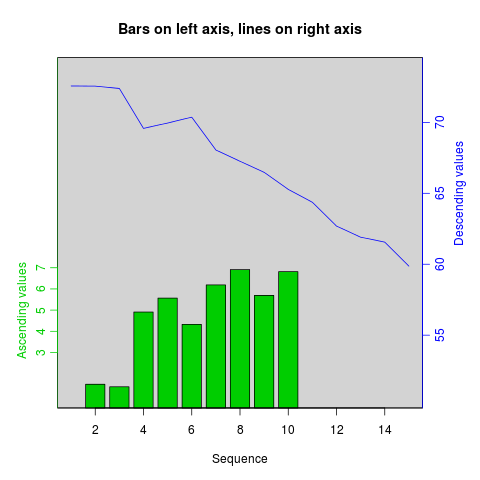
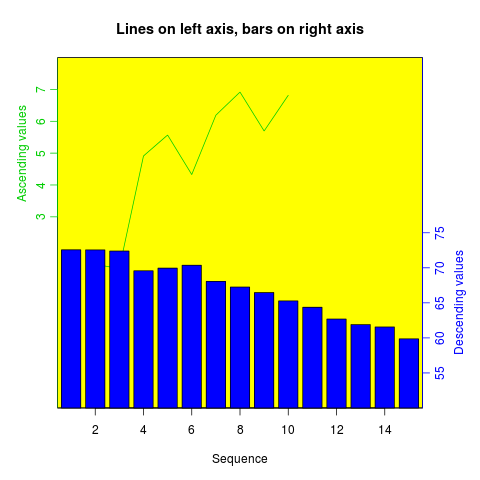
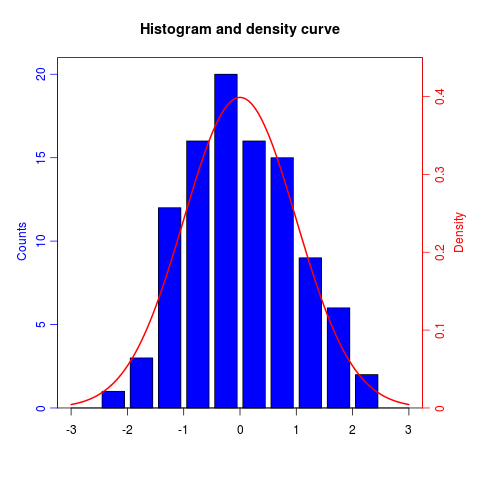
Jak sama nazwa wskazuje, twoord.plot() W plotrix działki pakietu z dwoma osiami porządkowymi.
library(plotrix)
example(twoord.plot)
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2014-09-06 00:30:56
Jedną z opcji jest zrobienie dwóch działek obok siebie. ggplot2 zapewnia ładną opcję dla tego z facet_wrap():
dat <- data.frame(x = c(rnorm(100), rnorm(100, 10, 2))
, y = c(rnorm(100), rlnorm(100, 9, 2))
, index = rep(1:2, each = 100)
)
require(ggplot2)
ggplot(dat, aes(x,y)) +
geom_point() +
facet_wrap(~ index, scales = "free_y")
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2011-05-26 18:00:56
To FAQ. Oto jedno starsze rozwiązanie, które podałem prawie 6 lat temu do galerii R Graph
Możesz spojrzeć np. na funkcję plotVolumeBars(), która łączy skalę absolutną i względną w jednym wykresie.
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2013-03-20 15:42:19
Jeśli można zrezygnować z wag / etykiet osi, można przeskalować dane do interwału (0, 1). Działa to na przykład dla różnych Traków "poruszających się" na chromosomach, gdy ogólnie interesuje Cię lokalna korelacja między ścieżkami i mają one różne skale (zasięg w tysiącach, Fst 0-1).
# rescale numeric vector into (0, 1) interval
# clip everything outside the range
rescale <- function(vec, lims=range(vec), clip=c(0, 1)) {
# find the coeficients of transforming linear equation
# that maps the lims range to (0, 1)
slope <- (1 - 0) / (lims[2] - lims[1])
intercept <- - slope * lims[1]
xformed <- slope * vec + intercept
# do the clipping
xformed[xformed < 0] <- clip[1]
xformed[xformed > 1] <- clip[2]
xformed
}
Wtedy, mając ramkę danych z chrom, position, coverage i fst kolumny, możesz zrobić coś takiego:
ggplot(d, aes(position)) +
geom_line(aes(y = rescale(fst))) +
geom_line(aes(y = rescale(coverage))) +
facet_wrap(~chrom)
Zaletą tego jest to, że nie jesteś ograniczony do dwóch trakcs.
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2015-04-02 20:15:49
Ja też proponuję twoord.stackplot() w pakiecie plotrix z większą liczbą dwóch osi porządkowych.
data<-read.table(text=
"e0AL fxAL e0CO fxCO e0BR fxBR anos
51.8 5.9 50.6 6.8 51.0 6.2 1955
54.7 5.9 55.2 6.8 53.5 6.2 1960
57.1 6.0 57.9 6.8 55.9 6.2 1965
59.1 5.6 60.1 6.2 57.9 5.4 1970
61.2 5.1 61.8 5.0 59.8 4.7 1975
63.4 4.5 64.0 4.3 61.8 4.3 1980
65.4 3.9 66.9 3.7 63.5 3.8 1985
67.3 3.4 68.0 3.2 65.5 3.1 1990
69.1 3.0 68.7 3.0 67.5 2.6 1995
70.9 2.8 70.3 2.8 69.5 2.5 2000
72.4 2.5 71.7 2.6 71.1 2.3 2005
73.3 2.3 72.9 2.5 72.1 1.9 2010
74.3 2.2 73.8 2.4 73.2 1.8 2015
75.2 2.0 74.6 2.3 74.2 1.7 2020
76.0 2.0 75.4 2.2 75.2 1.6 2025
76.8 1.9 76.2 2.1 76.1 1.6 2030
77.6 1.9 76.9 2.1 77.1 1.6 2035
78.4 1.9 77.6 2.0 77.9 1.7 2040
79.1 1.8 78.3 1.9 78.7 1.7 2045
79.8 1.8 79.0 1.9 79.5 1.7 2050
80.5 1.8 79.7 1.9 80.3 1.7 2055
81.1 1.8 80.3 1.8 80.9 1.8 2060
81.7 1.8 80.9 1.8 81.6 1.8 2065
82.3 1.8 81.4 1.8 82.2 1.8 2070
82.8 1.8 82.0 1.7 82.8 1.8 2075
83.3 1.8 82.5 1.7 83.4 1.9 2080
83.8 1.8 83.0 1.7 83.9 1.9 2085
84.3 1.9 83.5 1.8 84.4 1.9 2090
84.7 1.9 83.9 1.8 84.9 1.9 2095
85.1 1.9 84.3 1.8 85.4 1.9 2100", header=T)
require(plotrix)
twoord.stackplot(lx=data$anos, rx=data$anos,
ldata=cbind(data$e0AL, data$e0BR, data$e0CO),
rdata=cbind(data$fxAL, data$fxBR, data$fxCO),
lcol=c("black","red", "blue"),
rcol=c("black","red", "blue"),
ltype=c("l","o","b"),
rtype=c("l","o","b"),
lylab="Años de Vida", rylab="Hijos x Mujer",
xlab="Tiempo",
main="Mortalidad/Fecundidad:1950–2100",
border="grey80")
legend("bottomright", c(paste("Proy:",
c("A. Latina", "Brasil", "Colombia"))), cex=1,
col=c("black","red", "blue"), lwd=2, bty="n",
lty=c(1,1,2), pch=c(NA,1,1) )
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2015-07-29 12:31:48