Wielowymiarowa interpolacja spline w Pythonie / scipy?
Czy istnieje moduł biblioteki lub inny prosty sposób implementacji wielowymiarowej interpolacji spline w Pythonie?
W szczególności, mam zbiór danych skalarnych na regularnej trójwymiarowej siatce, którą muszę interpolować w niewielkiej liczbie punktów rozrzuconych po całej domenie. Od dwóch wymiarów używam scipy.interpolować.RectBivariateSpline , A ja zasadniczo Szukam rozszerzenia tego na dane trójwymiarowe.
The N-wymiarowe procedury interpolacji, które odkryłem, nie są wystarczająco dobre: wolałbym spliny niż LinearNDInterpolator dla gładkości, a mam zbyt wiele punktów danych (często ponad milion), aby np. radialna funkcja bazowa działała.
Jeśli ktoś zna bibliotekę Pythona, która może to zrobić, lub może jedną w innym języku, który mógłbym nazwać lub portować, byłbym naprawdę wdzięczny.
2 answers
Jeśli dobrze rozumiem twoje pytanie, Twoje wejściowe dane "obserwacji" są regularnie gridowane?
Jeśli tak, scipy.ndimage.map_coordinates robi dokładnie to, co chcesz.
Jest to trochę trudne do zrozumienia na pierwszym przejściu, ale zasadniczo, po prostu karmisz go sekwencją współrzędnych, które chcesz interpolować wartości siatki w Pixel/voxel/n-wymiarowej-Index współrzędnych.
Jako przykład 2D:
import numpy as np
from scipy import ndimage
import matplotlib.pyplot as plt
# Note that the output interpolated coords will be the same dtype as your input
# data. If we have an array of ints, and we want floating point precision in
# the output interpolated points, we need to cast the array as floats
data = np.arange(40).reshape((8,5)).astype(np.float)
# I'm writing these as row, column pairs for clarity...
coords = np.array([[1.2, 3.5], [6.7, 2.5], [7.9, 3.5], [3.5, 3.5]])
# However, map_coordinates expects the transpose of this
coords = coords.T
# The "mode" kwarg here just controls how the boundaries are treated
# mode='nearest' is _not_ nearest neighbor interpolation, it just uses the
# value of the nearest cell if the point lies outside the grid. The default is
# to treat the values outside the grid as zero, which can cause some edge
# effects if you're interpolating points near the edge
# The "order" kwarg controls the order of the splines used. The default is
# cubic splines, order=3
zi = ndimage.map_coordinates(data, coords, order=3, mode='nearest')
row, column = coords
nrows, ncols = data.shape
im = plt.imshow(data, interpolation='nearest', extent=[0, ncols, nrows, 0])
plt.colorbar(im)
plt.scatter(column, row, c=zi, vmin=data.min(), vmax=data.max())
for r, c, z in zip(row, column, zi):
plt.annotate('%0.3f' % z, (c,r), xytext=(-10,10), textcoords='offset points',
arrowprops=dict(arrowstyle='->'), ha='right')
plt.show()
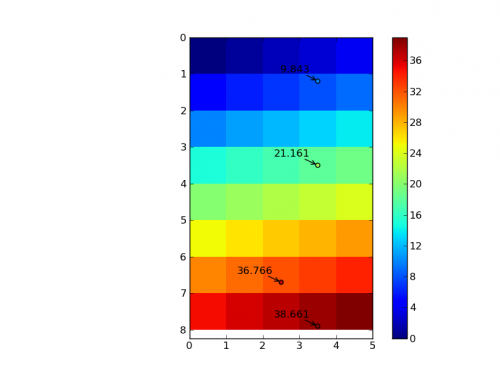
Aby to zrobić w n-wymiarach, musimy tylko przekazać w odpowiedniej wielkości tablicach:
import numpy as np
from scipy import ndimage
data = np.arange(3*5*9).reshape((3,5,9)).astype(np.float)
coords = np.array([[1.2, 3.5, 7.8], [0.5, 0.5, 6.8]])
zi = ndimage.map_coordinates(data, coords.T)
Jeśli chodzi o skalowanie i zużycie pamięci, map_coordinates utworzy przefiltrowaną kopię tablicy, jeśli używasz kolejności > 1 (tzn. nie liniowej interpolacji). Jeśli po prostu chcesz interpolować w bardzo małej liczbie punktów, jest to dość duży narzut. Nie zwiększa się jednak wraz z liczbą punktów, w których chcesz interpolować. Dopóki masz wystarczającą ilość pamięci RAM na jedną tymczasową kopię wejściowej macierzy danych, wszystko będzie dobrze.
Jeśli nie można przechowywać kopii danych w pamięci, można albo a) podać prefilter=False i order=1 i użyć interpolacji liniowej, albo b) zastąpić oryginalne dane wersją filtrowaną za pomocą ndimage.spline_filter, a następnie wywołać map_coordinates za pomocą prefilter=False.
Nawet jeśli masz wystarczającą ilość pamięci ram, utrzymywanie przefiltrowanego zestawu danych może być dużym przyspieszeniem, jeśli musisz wielokrotnie wywoływać map_coordinates (np. interaktywne użycie, itp.).
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2011-06-04 19:48:09
Płynna interpolacja splajnu w dim > 2 jest trudna do zaimplementowania, a więc nie ma zbyt wielu darmowych bibliotek, które mogłyby to zrobić (właściwie nie znam żadnej).
Możesz spróbować interpolacji z odwróconą odległością, Zobacz: Interpolacja z odwróconą odległością (IDW) z Pythonem . Powinno to przynieść dość płynne wyniki i skalować się lepiej niż RBF do większych zbiorów danych.
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2017-05-23 12:34:02