Efektywna metoda obliczania gęstości punktów o nieregularnym rozstawie
Próbuję wygenerować obrazy nakładek map, które pomogą w identyfikacji hot-spotów, czyli obszarów na mapie, które mają dużą gęstość punktów danych. Żaden z podejść, które próbowałem są wystarczająco szybkie dla moich potrzeb. Uwaga: zapomniałem wspomnieć, że algorytm powinien działać dobrze zarówno w scenariuszach niskiego i wysokiego zoomu (lub niskiej i wysokiej gęstości punktów danych).
Przejrzałem biblioteki numpy, pyplot i scipy, i najbliżej znalazłem numpy.histogram2d. jak można zobacz na poniższym obrazku, wyjście histogram2d jest raczej prymitywne. (Każdy obraz zawiera punkty nakładające mapę cieplną dla lepszego zrozumienia)
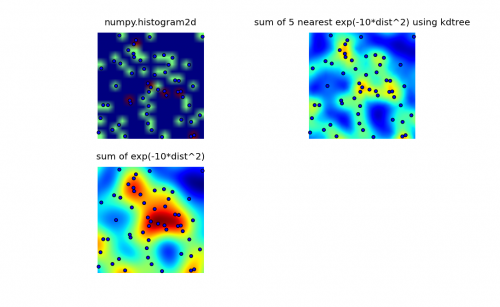 Moja druga próba polegała na iteracji wszystkich punktów danych, a następnie obliczeniu wartości hot-spot jako funkcji odległości. To dało lepiej wyglądający obraz, jednak jest zbyt wolny, aby używać go w mojej aplikacji. Ponieważ jest O (n), To Działa ok z 100 punktów, ale wieje, gdy używam mojego rzeczywistego zbioru danych z 30000 punktów.
Moja druga próba polegała na iteracji wszystkich punktów danych, a następnie obliczeniu wartości hot-spot jako funkcji odległości. To dało lepiej wyglądający obraz, jednak jest zbyt wolny, aby używać go w mojej aplikacji. Ponieważ jest O (n), To Działa ok z 100 punktów, ale wieje, gdy używam mojego rzeczywistego zbioru danych z 30000 punktów.
Mój finał próba polegała na zapisaniu danych w KDTree i wykorzystaniu najbliższych 5 punktów do obliczenia wartości hot-spot. Algorytm ten jest O(1), o wiele szybszy przy dużym zbiorze danych. To wciąż nie jest wystarczająco szybki, To Trwa około 20 sekund, aby wygenerować bitmapę 256x256, i chciałbym, aby to się stało w około 1 drugi raz.
Edit
Rozwiązanie do wygładzania boxsum dostarczone przez 6502 działa dobrze na wszystkich poziomach zoomu i jest znacznie szybsze niż moje oryginalne metody.
Filtr Gaussa rozwiązanie zaproponowane przez Luke ' a i Neila G jest najszybsze.
Możesz zobaczyć wszystkie cztery podejścia poniżej, używając w sumie 1000 punktów danych, przy 3-krotnym zoomie widać około 60 punktów.
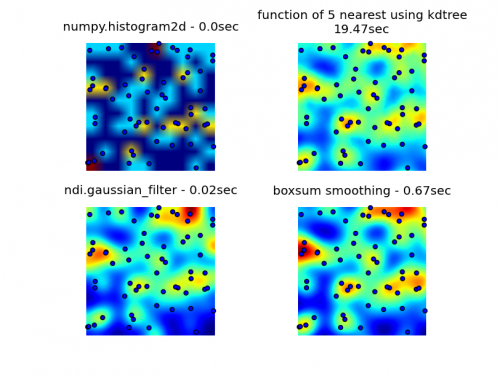
Kompletny kod, który generuje moje oryginalne 3 próby, rozwiązanie wygładzania boxsum dostarczone przez 6502 i filtr gaussowski sugerowany przez Luke ' a (ulepszony, aby lepiej obsługiwać krawędzie i umożliwić powiększanie) jest tutaj: {]}
import matplotlib
import numpy as np
from matplotlib.mlab import griddata
import matplotlib.cm as cm
import matplotlib.pyplot as plt
import math
from scipy.spatial import KDTree
import time
import scipy.ndimage as ndi
def grid_density_kdtree(xl, yl, xi, yi, dfactor):
zz = np.empty([len(xi),len(yi)], dtype=np.uint8)
zipped = zip(xl, yl)
kdtree = KDTree(zipped)
for xci in range(0, len(xi)):
xc = xi[xci]
for yci in range(0, len(yi)):
yc = yi[yci]
density = 0.
retvalset = kdtree.query((xc,yc), k=5)
for dist in retvalset[0]:
density = density + math.exp(-dfactor * pow(dist, 2)) / 5
zz[yci][xci] = min(density, 1.0) * 255
return zz
def grid_density(xl, yl, xi, yi):
ximin, ximax = min(xi), max(xi)
yimin, yimax = min(yi), max(yi)
xxi,yyi = np.meshgrid(xi,yi)
#zz = np.empty_like(xxi)
zz = np.empty([len(xi),len(yi)])
for xci in range(0, len(xi)):
xc = xi[xci]
for yci in range(0, len(yi)):
yc = yi[yci]
density = 0.
for i in range(0,len(xl)):
xd = math.fabs(xl[i] - xc)
yd = math.fabs(yl[i] - yc)
if xd < 1 and yd < 1:
dist = math.sqrt(math.pow(xd, 2) + math.pow(yd, 2))
density = density + math.exp(-5.0 * pow(dist, 2))
zz[yci][xci] = density
return zz
def boxsum(img, w, h, r):
st = [0] * (w+1) * (h+1)
for x in xrange(w):
st[x+1] = st[x] + img[x]
for y in xrange(h):
st[(y+1)*(w+1)] = st[y*(w+1)] + img[y*w]
for x in xrange(w):
st[(y+1)*(w+1)+(x+1)] = st[(y+1)*(w+1)+x] + st[y*(w+1)+(x+1)] - st[y*(w+1)+x] + img[y*w+x]
for y in xrange(h):
y0 = max(0, y - r)
y1 = min(h, y + r + 1)
for x in xrange(w):
x0 = max(0, x - r)
x1 = min(w, x + r + 1)
img[y*w+x] = st[y0*(w+1)+x0] + st[y1*(w+1)+x1] - st[y1*(w+1)+x0] - st[y0*(w+1)+x1]
def grid_density_boxsum(x0, y0, x1, y1, w, h, data):
kx = (w - 1) / (x1 - x0)
ky = (h - 1) / (y1 - y0)
r = 15
border = r * 2
imgw = (w + 2 * border)
imgh = (h + 2 * border)
img = [0] * (imgw * imgh)
for x, y in data:
ix = int((x - x0) * kx) + border
iy = int((y - y0) * ky) + border
if 0 <= ix < imgw and 0 <= iy < imgh:
img[iy * imgw + ix] += 1
for p in xrange(4):
boxsum(img, imgw, imgh, r)
a = np.array(img).reshape(imgh,imgw)
b = a[border:(border+h),border:(border+w)]
return b
def grid_density_gaussian_filter(x0, y0, x1, y1, w, h, data):
kx = (w - 1) / (x1 - x0)
ky = (h - 1) / (y1 - y0)
r = 20
border = r
imgw = (w + 2 * border)
imgh = (h + 2 * border)
img = np.zeros((imgh,imgw))
for x, y in data:
ix = int((x - x0) * kx) + border
iy = int((y - y0) * ky) + border
if 0 <= ix < imgw and 0 <= iy < imgh:
img[iy][ix] += 1
return ndi.gaussian_filter(img, (r,r)) ## gaussian convolution
def generate_graph():
n = 1000
# data points range
data_ymin = -2.
data_ymax = 2.
data_xmin = -2.
data_xmax = 2.
# view area range
view_ymin = -.5
view_ymax = .5
view_xmin = -.5
view_xmax = .5
# generate data
xl = np.random.uniform(data_xmin, data_xmax, n)
yl = np.random.uniform(data_ymin, data_ymax, n)
zl = np.random.uniform(0, 1, n)
# get visible data points
xlvis = []
ylvis = []
for i in range(0,len(xl)):
if view_xmin < xl[i] < view_xmax and view_ymin < yl[i] < view_ymax:
xlvis.append(xl[i])
ylvis.append(yl[i])
fig = plt.figure()
# plot histogram
plt1 = fig.add_subplot(221)
plt1.set_axis_off()
t0 = time.clock()
zd, xe, ye = np.histogram2d(yl, xl, bins=10, range=[[view_ymin, view_ymax],[view_xmin, view_xmax]], normed=True)
plt.title('numpy.histogram2d - '+str(time.clock()-t0)+"sec")
plt.imshow(zd, origin='lower', extent=[view_xmin, view_xmax, view_ymin, view_ymax])
plt.scatter(xlvis, ylvis)
# plot density calculated with kdtree
plt2 = fig.add_subplot(222)
plt2.set_axis_off()
xi = np.linspace(view_xmin, view_xmax, 256)
yi = np.linspace(view_ymin, view_ymax, 256)
t0 = time.clock()
zd = grid_density_kdtree(xl, yl, xi, yi, 70)
plt.title('function of 5 nearest using kdtree\n'+str(time.clock()-t0)+"sec")
cmap=cm.jet
A = (cmap(zd/256.0)*255).astype(np.uint8)
#A[:,:,3] = zd
plt.imshow(A , origin='lower', extent=[view_xmin, view_xmax, view_ymin, view_ymax])
plt.scatter(xlvis, ylvis)
# gaussian filter
plt3 = fig.add_subplot(223)
plt3.set_axis_off()
t0 = time.clock()
zd = grid_density_gaussian_filter(view_xmin, view_ymin, view_xmax, view_ymax, 256, 256, zip(xl, yl))
plt.title('ndi.gaussian_filter - '+str(time.clock()-t0)+"sec")
plt.imshow(zd , origin='lower', extent=[view_xmin, view_xmax, view_ymin, view_ymax])
plt.scatter(xlvis, ylvis)
# boxsum smoothing
plt3 = fig.add_subplot(224)
plt3.set_axis_off()
t0 = time.clock()
zd = grid_density_boxsum(view_xmin, view_ymin, view_xmax, view_ymax, 256, 256, zip(xl, yl))
plt.title('boxsum smoothing - '+str(time.clock()-t0)+"sec")
plt.imshow(zd, origin='lower', extent=[view_xmin, view_xmax, view_ymin, view_ymax])
plt.scatter(xlvis, ylvis)
if __name__=='__main__':
generate_graph()
plt.show()
6 answers
Takie podejście jest podobne do kilku poprzednich odpowiedzi: zwiększ Piksel dla każdego miejsca, a następnie wygładź obraz filtrem Gaussa. Obraz 256x256 działa w około 350ms na moim 6-letnim laptopie.
import numpy as np
import scipy.ndimage as ndi
data = np.random.rand(30000,2) ## create random dataset
inds = (data * 255).astype('uint') ## convert to indices
img = np.zeros((256,256)) ## blank image
for i in xrange(data.shape[0]): ## draw pixels
img[inds[i,0], inds[i,1]] += 1
img = ndi.gaussian_filter(img, (10,10))
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2017-05-08 22:00:59
Bardzo prostą implementacją, która może być wykonana (z C) w czasie rzeczywistym i która zajmuje tylko ułamki sekundy w czystym Pythonie jest po prostu obliczyć wynik w przestrzeni ekranu.
Algorytm jest
- Przydziel macierz końcową (np. 256x256) ze wszystkimi zerami
- dla każdego punktu w zbiorze danych przyrost odpowiedniej komórki
- Zastąp każdą komórkę macierzy sumą wartości macierzy w polu NXN wyśrodkowanym na komórce. Powtórz ten krok kilka razy.
- Scale result and output
Obliczenie sumy pola może być wykonane bardzo szybko i niezależnie od N za pomocą tabeli sumy. Każde obliczenie wymaga tylko dwóch skanów matrycy... złożoność całkowita to O (S + WHP), Gdzie S to liczba punktów; W, H to szerokość i wysokość wyjścia, A P to liczba przejść wygładzających.
Poniżej znajduje się kod dla czystej implementacji Pythona( również bardzo Nie zoptymalizowanej); z 30000 punktów i wyjściem 256x256 obraz w skali szarości obliczenie wynosi 0,5 S, w tym skalowanie liniowe do 0..255 i oszczędzanie a .plik pgm (N = 5, 4 przejścia).
def boxsum(img, w, h, r):
st = [0] * (w+1) * (h+1)
for x in xrange(w):
st[x+1] = st[x] + img[x]
for y in xrange(h):
st[(y+1)*(w+1)] = st[y*(w+1)] + img[y*w]
for x in xrange(w):
st[(y+1)*(w+1)+(x+1)] = st[(y+1)*(w+1)+x] + st[y*(w+1)+(x+1)] - st[y*(w+1)+x] + img[y*w+x]
for y in xrange(h):
y0 = max(0, y - r)
y1 = min(h, y + r + 1)
for x in xrange(w):
x0 = max(0, x - r)
x1 = min(w, x + r + 1)
img[y*w+x] = st[y0*(w+1)+x0] + st[y1*(w+1)+x1] - st[y1*(w+1)+x0] - st[y0*(w+1)+x1]
def saveGraph(w, h, data):
X = [x for x, y in data]
Y = [y for x, y in data]
x0, y0, x1, y1 = min(X), min(Y), max(X), max(Y)
kx = (w - 1) / (x1 - x0)
ky = (h - 1) / (y1 - y0)
img = [0] * (w * h)
for x, y in data:
ix = int((x - x0) * kx)
iy = int((y - y0) * ky)
img[iy * w + ix] += 1
for p in xrange(4):
boxsum(img, w, h, 2)
mx = max(img)
k = 255.0 / mx
out = open("result.pgm", "wb")
out.write("P5\n%i %i 255\n" % (w, h))
out.write("".join(map(chr, [int(v*k) for v in img])))
out.close()
import random
data = [(random.random(), random.random())
for i in xrange(30000)]
saveGraph(256, 256, data)
Edit
Oczywiście sama definicja gęstości w Twoim przypadku zależy od promienia rozdzielczości, czy gęstość jest po prostu +inf, gdy trafisz w punkt, a zero, gdy nie?
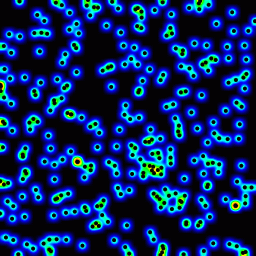
Poniżej znajduje się animacja zbudowana za pomocą powyższego programu z kilkoma kosmetycznymi zmianami:
- używane
sqrt(average of squared values)zamiastsumdo uśredniania pass - kodowanie kolorami wyników
- rozciąganie wyniku, aby zawsze używać pełnej skali kolorów
- rysowane antyaliasowane czarne kropki, gdzie punkty danych są
- wykonano animację zwiększając promień z 2 do 40
Całkowity czas obliczeniowy 39 klatek poniższej animacji w tej kosmetycznej wersji wynosi 5,4 sekundy dla PyPy i 26 sekund dla standardowego Pythona.
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2011-07-12 09:04:37
Histogramy
Sposób histogramu nie jest najszybszy i nie może odróżnić arbitralnie małego rozdziału punktów od 2 * sqrt(2) * b (gdzie b jest szerokością bin).
Nawet jeśli konstruujesz pojemniki x i y osobno(O (N)), nadal musisz wykonać splot ab (liczba pojemników w każdą stronę), który jest bliski N^2 dla dowolnego gęstego systemu, a nawet większy dla rzadkiego (cóż, ab >> N^2 w rzadkim systemie.)
Patrząc na powyższy kod, wydaje się, że masz pętlę w grid_density(), która przebiega przez liczbę pojemników W y wewnątrz pętli o liczbie pojemników W x, dlatego otrzymujesz wydajność O (N^2) (chociaż jeśli już masz kolejność N, którą powinieneś narysować na różnych liczbach elementów, aby zobaczyć, to po prostu będziesz musiał uruchomić mniej kodu na cykl).
Jeśli chcesz mieć rzeczywistą funkcję odległości, musisz zacząć szukać algorytmów wykrywania kontaktu.
Kontakt Detekcja
Naiwne algorytmy wykrywania kontaktu pojawiają się w O (N^2) w czasie RAM lub CPU, ale istnieje algorytm, słusznie lub niesłusznie przypisywany Munjizie w St.Mary ' s college London, który działa w czasie liniowym i RAM.
Możesz o tym przeczytać i zaimplementować to samodzielnie z jego książki , Jeśli chcesz.
Sam napisałem ten kod, w rzeczywistości
Napisałem implementację C w Pythonie w 2D, która nie jest naprawdę gotowy do produkcji (nadal jest jednowątkowy, itp.), ale będzie działał tak blisko O(N), Jak pozwoli na to zestaw danych. Ustawiasz "Rozmiar elementu", który działa jako rozmiar bin (kod wywoła interakcje na wszystko w b innego punktu, a czasami między b i 2 * sqrt(2) * b), nadajesz mu tablicę (natywną listę Pythona) obiektów z właściwością x i y, a my C moduł oddzwoni do wybranej przez Ciebie funkcji Pythona, aby uruchomić funkcję interakcji dla dopasowanych par obiektów. żywioły. jest przeznaczony do prowadzenia symulacji siły nacisku, ale będzie działał dobrze również na ten problem.
Ponieważ jeszcze go nie wydałem, ponieważ pozostałe bity biblioteki nie są jeszcze gotowe, będę musiał podać zip mojego obecnego źródła, ale część wykrywania kontaktu jest solidna. Kod jest LGPL ' D.
Będziesz potrzebował Cythona i kompilatora c, aby to działało, a został przetestowany i działa tylko pod * Nix environemnts, jeśli jesteś na windows będziesz potrzebował MinGW c kompilator dla Cythona w ogóle działa .
Po zainstalowaniu Cythona, budowanie /instalowanie pynet powinno być przypadkiem uruchomienia setup.py.
Interesującą Cię funkcją jest pynet.d2.run_contact_detection(py_elements, py_interaction_function, py_simulation_parameters) (i powinieneś sprawdzić element classes I SimulationParameters na tym samym poziomie, jeśli chcesz, aby rzucała mniej błędów - zajrzyj do pliku w archive-root/pynet/d2/__init__.py, aby zobaczyć implementacje klas, są to trywialne posiadacze danych z przydatnymi konstruktorami.)
(zaktualizuję tę odpowiedź o public mercurial repo, gdy kod jest gotowy do bardziej ogólnego wydania...)
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2011-07-11 16:29:04
Twoje rozwiązanie jest w porządku, ale jednym z oczywistych problemów jest to, że dostajesz ciemne obszary, mimo że w środku jest punkt.
Zamiast tego wyśrodkowałbym N-wymiarowy Gaussian na każdym punkcie i oszacowałbym sumę nad każdym punktem, który chcesz wyświetlić. Aby zredukować go do czasu liniowego w powszechnym przypadku, użyj query_ball_point, aby rozważyć tylko punkty w obrębie kilku odchyleń standardowych.
Jeśli okaże się, że KDTree jest naprawdę powolny, dlaczego nie wywołać query_ball_point raz na pięć pikseli z nieco większy próg? Nie zaszkodzi ocenić zbyt wielu Gaussów.
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2011-07-11 16:56:07
Można to zrobić za pomocą dwuwymiarowego, rozdzielnego splotu (scipy.ndimage.convolve1d) oryginalnego obrazu z jądrem w kształcie Gaussa. Przy rozmiarze obrazu MxM i rozmiarze filtra P, złożoność wynosi O (PM^2) przy użyciu filtrowania rozdzielnego. Złożoność "Big-Oh" jest bez wątpienia większa, ale możesz skorzystać z wydajnych operacji numpy, które powinny znacznie przyspieszyć twoje obliczenia.
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2011-07-11 19:12:11
Tylko uwaga, histogram2d funkcja powinna działać dobrze do tego. Bawiłeś się różnymi pojemnikami? Twój początkowy histogram2d Wykres wydaje się używać tylko domyślnych rozmiarów bin... ale nie ma powodu, aby oczekiwać, że domyślne rozmiary dadzą Ci pożądaną reprezentację. Mimo to wiele innych rozwiązań jest imponujących.
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2012-08-04 15:19:30