Dopasowanie krzywej gęstości do histogramu w R
Czy istnieje funkcja w R, która pasuje do krzywej do histogramu?
Załóżmy, że masz następujący histogram
hist(c(rep(65, times=5), rep(25, times=5), rep(35, times=10), rep(45, times=4)))
To pytanie jest raczej podstawowe, ale nie mogę znaleźć odpowiedzi na R w Internecie.
5 answers
Jeśli dobrze rozumiem twoje pytanie, prawdopodobnie chcesz oszacować gęstość wraz z histogramem:
X <- c(rep(65, times=5), rep(25, times=5), rep(35, times=10), rep(45, times=4))
hist(X, prob=TRUE) # prob=TRUE for probabilities not counts
lines(density(X)) # add a density estimate with defaults
lines(density(X, adjust=2), lty="dotted") # add another "smoother" density
Edit a long while later:
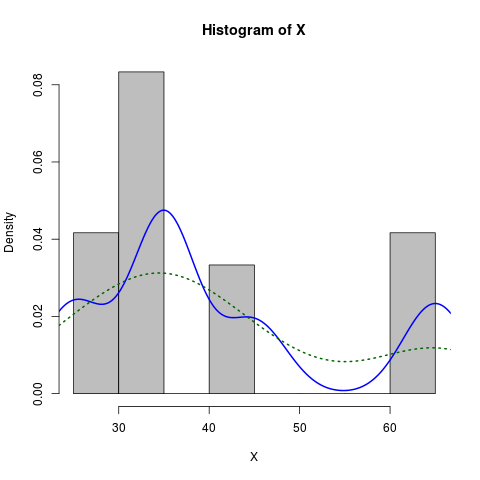
Oto nieco bardziej ubrana wersja:
X <- c(rep(65, times=5), rep(25, times=5), rep(35, times=10), rep(45, times=4))
hist(X, prob=TRUE, col="grey")# prob=TRUE for probabilities not counts
lines(density(X), col="blue", lwd=2) # add a density estimate with defaults
lines(density(X, adjust=2), lty="dotted", col="darkgreen", lwd=2)
Wraz z wykresem, który tworzy:
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2014-05-04 18:33:03
Takie rzeczy są łatwe z ggplot2
library(ggplot2)
dataset <- data.frame(X = c(rep(65, times=5), rep(25, times=5),
rep(35, times=10), rep(45, times=4)))
ggplot(dataset, aes(x = X)) +
geom_histogram(aes(y = ..density..)) +
geom_density()
Lub naśladować wynik rozwiązania Dirka
ggplot(dataset, aes(x = X)) +
geom_histogram(aes(y = ..density..), binwidth = 5) +
geom_density()
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2018-06-03 12:06:06
Oto Jak to robię:
foo <- rnorm(100, mean=1, sd=2)
hist(foo, prob=TRUE)
curve(dnorm(x, mean=mean(foo), sd=sd(foo)), add=TRUE)
Dodatkowym ćwiczeniem jest zrobienie tego z pakietem ggplot2 ...
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2012-05-04 12:06:19
Dirk wyjaśnił, jak wykreślić funkcję gęstości na histogramie. Ale czasami możesz chcieć przejść z mocniejszym założeniem przekrzywionego rozkładu normalnego i wykreślić, że zamiast gęstości. Możesz oszacować parametry rozkładu i wykreślić go za pomocą pakietu sn :
> sn.mle(y=c(rep(65, times=5), rep(25, times=5), rep(35, times=10), rep(45, times=4)))
$call
sn.mle(y = c(rep(65, times = 5), rep(25, times = 5), rep(35,
times = 10), rep(45, times = 4)))
$cp
mean s.d. skewness
41.46228 12.47892 0.99527
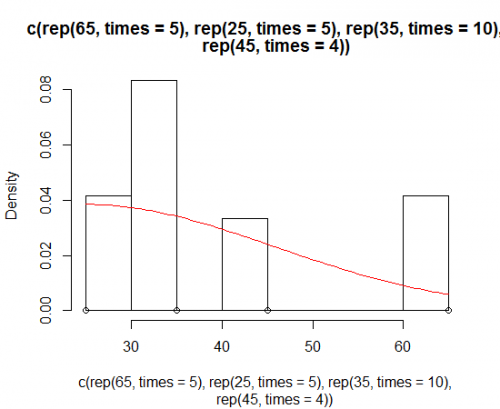
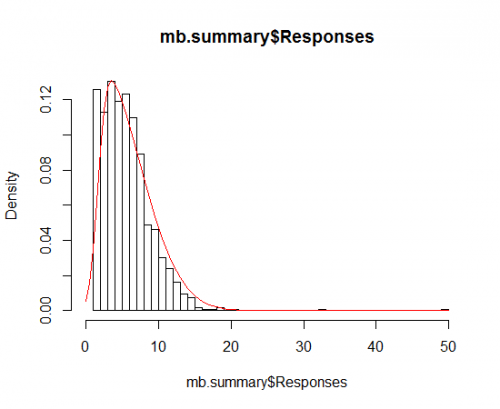
To prawdopodobnie działa lepiej na danych, które są bardziej Przekrzywione-normalne:
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2017-05-23 12:26:26
Miałem ten sam problem, ale rozwiązanie Dirka nie zadziałało. Za każdym razem otrzymywałem ten Komunikat ostrzegawczy
"prob" is not a graphical parameter
Przeczytałem ?hist i znalazłem około freq: a logical vector set TRUE by default.
Kod, który działał dla mnie to
hist(x,freq=FALSE)
lines(density(x),na.rm=TRUE)
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2019-02-17 16:44:35