Dopasowanie funkcji nieliniowej do danych/obserwacji za pomocą pyMCMC/pyMC
Próbuję dopasować niektóre dane za pomocą funkcji Gaussa (i bardziej złożonych). Poniżej stworzyłem mały przykład.
Moje pierwsze pytanie brzmi: Czy robię to dobrze?Moje drugie pytanie brzmi: Jak dodać błąd w kierunku x, czyli w pozycji X obserwacji / danych?
Bardzo trudno jest znaleźć fajne Poradniki, Jak zrobić tego rodzaju regresję w pyMC. Być może dlatego, że łatwiej jest korzystać z niektórych najmniej kwadratów, lub podobne podejście, I jednak mają wiele parametrów w końcu i trzeba zobaczyć, jak dobrze możemy je ograniczyć i porównać różne modele, pyMC wydawało się dobrym wyborem do tego.
import pymc
import numpy as np
import matplotlib.pyplot as plt; plt.ion()
x = np.arange(5,400,10)*1e3
# Parameters for gaussian
amp_true = 0.2
size_true = 1.8
ps_true = 0.1
# Gaussian function
gauss = lambda x,amp,size,ps: amp*np.exp(-1*(np.pi**2/(3600.*180.)*size*x)**2/(4.*np.log(2.)))+ps
f_true = gauss(x=x,amp=amp_true, size=size_true, ps=ps_true )
# add noise to the data points
noise = np.random.normal(size=len(x)) * .02
f = f_true + noise
f_error = np.ones_like(f_true)*0.05*f.max()
# define the model/function to be fitted.
def model(x, f):
amp = pymc.Uniform('amp', 0.05, 0.4, value= 0.15)
size = pymc.Uniform('size', 0.5, 2.5, value= 1.0)
ps = pymc.Normal('ps', 0.13, 40, value=0.15)
@pymc.deterministic(plot=False)
def gauss(x=x, amp=amp, size=size, ps=ps):
e = -1*(np.pi**2*size*x/(3600.*180.))**2/(4.*np.log(2.))
return amp*np.exp(e)+ps
y = pymc.Normal('y', mu=gauss, tau=1.0/f_error**2, value=f, observed=True)
return locals()
MDL = pymc.MCMC(model(x,f))
MDL.sample(1e4)
# extract and plot results
y_min = MDL.stats()['gauss']['quantiles'][2.5]
y_max = MDL.stats()['gauss']['quantiles'][97.5]
y_fit = MDL.stats()['gauss']['mean']
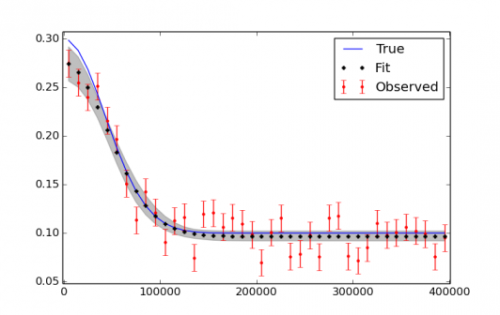
plt.plot(x,f_true,'b', marker='None', ls='-', lw=1, label='True')
plt.errorbar(x,f,yerr=f_error, color='r', marker='.', ls='None', label='Observed')
plt.plot(x,y_fit,'k', marker='+', ls='None', ms=5, mew=2, label='Fit')
plt.fill_between(x, y_min, y_max, color='0.5', alpha=0.5)
plt.legend()
Zdaję sobie sprawę, że być może będę musiał uruchomić więcej iteracji, użyć burn in i thinning w końcu. Rysunek przedstawiający dane i dopasowanie znajduje się poniżej.
2 answers
Moje pierwsze pytanie brzmi, Czy robię to dobrze?Tak! Musisz załączyć okres wypalenia, o którym wiesz. Lubię wyrzucać pierwszą połowę moich próbek. Nie musisz robić przerzedzania, ale czasami sprawi to, że Twoje post-MCMC będzie szybciej przetwarzać i mniejsze do przechowywania. Jedyne, co radzę, to ustawić losowe ziarno, aby wyniki były "powtarzalne":
np.random.seed(12345) da radę.
A gdybym naprawdę dawał za dużo Rada, powiedziałbym import seaborn, Aby matplotlib wyniki były trochę piękniejsze.
Moje drugie pytanie brzmi: jak dodać błąd w kierunku x, tzn. w pozycji X obserwacji / danych?
Jednym ze sposobów jest dołączenie zmiennej utajonej dla każdego błędu. To działa w twoim przykładzie, ale nie będzie możliwe, jeśli masz więcej obserwacji. Podam mały przykład na początek tej drogi:
# add noise to observed x values
x_obs = pm.rnormal(mu=x, tau=(1e4)**-2)
# define the model/function to be fitted.
def model(x_obs, f):
amp = pm.Uniform('amp', 0.05, 0.4, value= 0.15)
size = pm.Uniform('size', 0.5, 2.5, value= 1.0)
ps = pm.Normal('ps', 0.13, 40, value=0.15)
x_pred = pm.Normal('x', mu=x_obs, tau=(1e4)**-2) # this allows error in x_obs
@pm.deterministic(plot=False)
def gauss(x=x_pred, amp=amp, size=size, ps=ps):
e = -1*(np.pi**2*size*x/(3600.*180.))**2/(4.*np.log(2.))
return amp*np.exp(e)+ps
y = pm.Normal('y', mu=gauss, tau=1.0/f_error**2, value=f, observed=True)
return locals()
MDL = pm.MCMC(model(x_obs, f))
MDL.use_step_method(pm.AdaptiveMetropolis, MDL.x_pred) # use AdaptiveMetropolis to "learn" how to step
MDL.sample(200000, 100000, 10) # run chain longer since there are more dimensions
Wygląda na to, że może być trudno uzyskać dobre odpowiedzi jeśli masz hałas w x i y:
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2014-07-18 21:37:25
Przekonwertowałem powyższą odpowiedź Abrahama Flaxmana na PyMC3, na wypadek gdyby ktoś jej potrzebował. Niektóre bardzo drobne zmiany, ale mogą być mylące.
Pierwszy polega na tym, że dekorator deterministyczny @Deterministic jest zastępowany przez funkcję wywołania podobną do rozkładu var=pymc3.Deterministic(). Po drugie, podczas generowania wektora normalnie rozłożonych zmiennych losowych,
rvs = pymc2.rnormal(mu=mu, tau=tau)
Zastępuje się przez
rvs = pymc3.Normal('var_name', mu=mu, tau=tau,shape=size(var)).random()
Pełny kod jest następujący:
import numpy as np
from pymc3 import *
import matplotlib.pyplot as plt
# set random seed for reproducibility
np.random.seed(12345)
x = np.arange(5,400,10)*1e3
# Parameters for gaussian
amp_true = 0.2
size_true = 1.8
ps_true = 0.1
#Gaussian function
gauss = lambda x,amp,size,ps: amp*np.exp(-1*(np.pi**2/(3600.*180.)*size*x)**2/(4.*np.log(2.)))+ps
f_true = gauss(x=x,amp=amp_true, size=size_true, ps=ps_true )
# add noise to the data points
noise = np.random.normal(size=len(x)) * .02
f = f_true + noise
f_error = np.ones_like(f_true)*0.05*f.max()
with Model() as model3:
amp = Uniform('amp', 0.05, 0.4, testval= 0.15)
size = Uniform('size', 0.5, 2.5, testval= 1.0)
ps = Normal('ps', 0.13, 40, testval=0.15)
gauss=Deterministic('gauss',amp*np.exp(-1*(np.pi**2*size*x/(3600.*180.))**2/(4.*np.log(2.)))+ps)
y =Normal('y', mu=gauss, tau=1.0/f_error**2, observed=f)
start=find_MAP()
step=NUTS()
trace=sample(2000,start=start)
# extract and plot results
y_min = np.percentile(trace.gauss,2.5,axis=0)
y_max = np.percentile(trace.gauss,97.5,axis=0)
y_fit = np.percentile(trace.gauss,50,axis=0)
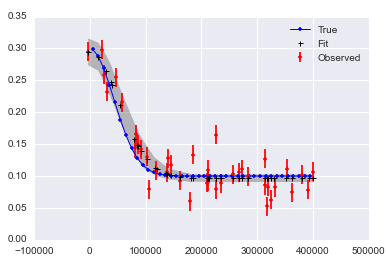
plt.plot(x,f_true,'b', marker='None', ls='-', lw=1, label='True')
plt.errorbar(x,f,yerr=f_error, color='r', marker='.', ls='None', label='Observed')
plt.plot(x,y_fit,'k', marker='+', ls='None', ms=5, mew=1, label='Fit')
plt.fill_between(x, y_min, y_max, color='0.5', alpha=0.5)
plt.legend()
Które wyniki w
Dla błędów w x (zwróć uwagę na przyrostek " x " do zmiennych):
# define the model/function to be fitted in PyMC3:
with Model() as modelx:
x_obsx = pm3.Normal('x_obsx',mu=x, tau=(1e4)**-2,shape=40).random()
ampx = Uniform('ampx', 0.05, 0.4, testval= 0.15)
sizex = Uniform('sizex', 0.5, 2.5, testval= 1.0)
psx = Normal('psx', 0.13, 40, testval=0.15)
x_pred = Normal('x_pred', mu=x_obsx, tau=(1e4)**-2*np.ones_like(x_obsx),testval=5*np.ones_like(x_obsx),shape=40) # this allows error in x_obs
gauss=Deterministic('gauss',ampx*np.exp(-1*(np.pi**2*sizex*x_pred/(3600.*180.))**2/(4.*np.log(2.)))+psx)
y = Normal('y', mu=gauss, tau=1.0/f_error**2, observed=f)
start=find_MAP()
step=NUTS()
tracex=sample(20000,start=start)
Co daje:
Ostatnią obserwacją jest to, że podczas wykonywania
traceplot(tracex[100:])
plt.tight_layout();
(wynik nie pokazany), widzimy, że sizex wydaje się cierpieć z powodu "tłumienia" lub "rozcieńczenia regresji" z powodu błędu w pomiarze x.
Warning: date(): Invalid date.timezone value 'Europe/Kyiv', we selected the timezone 'UTC' for now. in /var/www/agent_stack/data/www/doraprojects.net/template/agent.layouts/content.php on line 54
2016-02-10 16:29:09